Analyse génétique de la « fromageabilité » du lait de vache prédite par spectrométrie dans le moyen infrarouge en race Montbéliarde
Chapeau
La qualité fromagère (ou « fromageabilité ») d’un lait est un caractère économiquement important mais difficile à mesurer. Le projet From’MIR, conduit en race Montbéliarde et en zone AOP Comté, a montré que la spectrométrie dans le moyen infrarouge, utilisée en routine pour prédire la composition du lait, permet aussi de prédire sa « fromageabilité ». Il devient ainsi possible d’étudier le déterminisme génétique des aptitudes fromagères et de les améliorer par sélection.
Introduction
Le lait des ruminants, et en particulier celui des bovins, est utilisé pour fabriquer du fromage depuis les débuts de la domestication. L’ajout de présure, naturellement présente dans la caillette des veaux non sevrés, et/ou l’action de bactéries lactiques dans le lait entraînent sa coagulation, c’est-à-dire la formation d’un gel suivi de la séparation du caillé (ou fromage) du lactosérum (ou « petit lait »), et transforme ainsi une denrée liquide périssable en un fromage. Celui-ci est beaucoup plus facile à stocker et à conserver, et aussi beaucoup plus digeste, le lactose étant consommé par les bactéries lactiques lors de la transformation du lait ou évacué dans le lactosérum. Ainsi, aujourd’hui dans le monde, plus d’un tiers du lait produit par les vaches est transformé en fromage. La France, avec sa très grande diversité de fromages (1200 variétés), est un des premiers pays au monde pour la production, la consommation et l’exportation de fromages. Parmi les fromages français au lait de vache, 28 ont reçu une Appellation d’Origine Protégée (AOP) parmi lesquels le Comté, produit en Franche-Comté et premier fromage AOP de France (CNAOL, 2018).
Derrière les premières étapes de transformation du lait en fromage se cachent des mécanismes physicochimiques complexes liés à la composition du lait, notamment en protéines (Wedholm et al., 2006). En fonction de la composition du lait qu’elle produit, une vache peut donc fournir un lait plus ou moins « fromageable », i.e. plus ou moins apte à la transformation selon ses aptitudes d’acidification et de coagulation et ses rendements fromagers. Les caractères de composition ou de « fromageabilité » du lait sont difficiles à mesurer avec les méthodes de référence, soit lors du processus de fabrication, soit avec des tests de laboratoire mimant certaines phases de fabrication, ce qui rend impossible leur étude à grande échelle. La spectrométrie dans le moyen infrarouge (MIR) permet de prédire la composition de la matière organique en milieu liquide et en particulier celle du lait (De Marchi et al., 2014). Utilisée en routine dans le cadre du contrôle laitier pour prédire notamment les taux butyreux et protéique qui entrent dans la grille de paiement du lait, elle offre une alternative aux mesures de référence longues et coûteuses.
Le projet de recherche From’MIR a été mis en place dans le but d’exploiter la spectrométrie MIR pour prédire et étudier la « fromageabilité » (aptitudes physicochimiques) du lait en race Montbéliarde dans la zone AOP Comté. Un des volets du projet From’MIR est l’analyse génétique des aptitudes fromagères prédites à partir des spectres MIR. Nous faisons ici une synthèse de l’ensemble des résultats obtenus lors de l’estimation des paramètres génétiques, de la recherche de gènes et de variants causaux et enfin, de l’étude des possibilités de sélection des vaches pour qu’elles produisent un lait plus « fromageable ».
1. Prédiction de la « fromageabilité » du lait dans le projet From’MIR
La spectrométrie MIR du lait repose sur l’absorbance de la lumière infrarouge par les composants chimiques du lait. Les aptitudes fromagères d’un lait étant liées à sa composition chimique, il est possible de les prédire à partir d’un spectre MIR du lait. Grâce à une population de calibration, constituée de laits représentatifs de la variabilité de la population étudiée et analysés à la fois par spectrométrie MIR et par des méthodes de référence, et à des méthodes statistiques adaptées, il est possible de développer des équations de prédiction de la « fromageabilité » du lait. Une fois établies, ces équations peuvent ensuite être appliquées à tout spectre MIR pour prédire les aptitudes fromagères d’un lait (encadré 1).
Encadré 1. Comment prédit-on un phénotype à partir d’un spectre MIR ?
La spectrométrie dans le moyen infrarouge (MIR) repose sur l’interaction entre les composants chimiques de la matière organique (par exemple celle du lait) et la lumière infrarouge. Dans la gamme de longueurs d’ondes du MIR, les mouvements des molécules sont associés à une plus ou moins grande absorption de l’énergie fournie. La quantité d’énergie absorbée par la matière pour chaque longueur d’onde du spectre permet de caractériser les groupements fonctionnels, les liaisons chimiques et la structure des molécules qui constituent la matière organique.
Les aptitudes fromagères d’un lait étant liées à sa composition chimique, il est possible de les prédire à partir d’un spectre MIR du lait. Dans le projet From’MIR, 416 laits ont été analysés par spectrométrie MIR et par des mesures de référence des aptitudes fromagères. Cette population de calibration a été analysée par « Partial Least Squares » (PLS) pour établir les équations de prédiction qui ont ensuite été appliquées à plus de 6 millions de spectres MIR de 400 000 vaches de race Montbéliarde.
1.1. Population de calibration : échantillonnage et analyses de référence
Dans le projet From’MIR, 250 laits individuels, 100 laits de tanks de troupeaux et 70 laits de cuves fromagères de 55 fromageries ont été échantillonnés pour être représentatifs de la variabilité des aptitudes fromagères en région Franche-Comté. Les laits de mélange (tanks + cuves) ont été collectés en février-mars et mai-juin 2015 (deux saisons de vêlage) dans 100 troupeaux de tailles différentes, répartis sur toute la région et présentant des taux protéiques annuels variés lors de la campagne précédente. L’échantillonnage des laits individuels a été réalisé entre janvier et juin 2016 sur des vaches à différents stades de lactation qui présentaient une large variété de génotypes aux lactoprotéines (Fang et al., 2016) et avec moins de 106 cellules somatiques par mL et 105 UFC/mL. Sur les 250 échantillons de lait individuels recueillis, 99 provenaient de la traite du matin, 98 de la traite du soir et 53 étaient un mélange de laits prélevés en proportions relatives à la traite du matin et du soir.
Au total donc, 416 échantillons de lait frais, cru et entier ont été collectés par les techniciens des Entreprises de Conseil en Élevage (ECEL) dans des conditions strictes d’hygiène, immédiatement refroidis à 4°C et analysés dans les 24h. Chaque échantillon a été séparé en deux aliquotes. La première a été conservée avec du bronopol et analysée par spectrométrie MIR (MilkoScan FT6000, Foss Electric A/S, Hillerød, Danemark ; laboratoire d’analyse de l’ECEL25-90). La seconde a été utilisée pour mesurer les aptitudes fromagères avec les méthodes de référence en laboratoire (INRA et ENILBio de Poligny, Jura). Avant mesure des rendements et des paramètres de coagulation, le lait a été standardisé pour le pH par ajout d’acide lactique. Les trois rendements fromagers de laboratoire ont été mesurés en double selon la méthode de Hurtaud et al. . Les paramètres de coagulation et d’acidification ont été mesurés pour deux modèles types de technologie fromagère : Pâte Pressée Cuite (PPC) et pâte molle (PM). Chaque échantillon de lait a ainsi été coagulé par ajout de présure ou acidifié par ajout de bactéries lactiques selon deux protocoles différents. Les paramètres de coagulation ont été mesurés sur 10 mL de lait après 30 min à 32°C avec un instrument Formoptic (Foss Electric A/S, Hillerød, Danemark). La cinétique d’acidification a été enregistrée sur 100 mL de lait pendant 20h avec le système CINAC (Corrieu et al., 1988). Au total, 24 paramètres fromagers de rendement (3), coagulation (13) et acidification (8) ont été mesurés. Tous les détails peuvent être trouvés dans Sanchez et al. .
1.2. Équations de prédiction de la « fromageabilité » à partir des spectres MIR
Chaque spectre MIR étant constitué d’un grand nombre de variables (1060 longueurs d’onde, 446 après élimination des longueurs d’onde qui correspondent à l’absorption de l’eau), des méthodes statistiques adaptées doivent être appliquées pour établir les équations de prédiction. La méthode qui a donné les meilleurs résultats dans le projet From’MIR est la méthode de régression PLS « Partial Least Square » couplée à un algorithme de sélection des longueurs d’onde les plus informatives. Sur les 24 caractères fromagers mesurés, les neuf mieux prédits (El Jabri et al., 2019) ont été retenus pour la suite des analyses (tableau 1), ce qui correspond à des prédictions de qualité moyenne à excellente.
Tableau 1. Description des caractères fromagers et précision des équations de prédiction à partir des spectres MIR.
Noma |
Description |
R²VALb |
SEVALb |
|---|---|---|---|
Rendement (%) |
|||
RDT_FRAIS |
Frais : 100 x (g caillé / g lait)
|
0,86 |
2,8 |
RDT_ES |
En extrait sec : 100 x [1- (g ES lactosérum / g ES lait)] |
0,89 |
1,7 |
RDT_MSU |
En matière sèche utile : (TP + TB) x (g lait / g caillé) |
0,54 |
15,5 |
Coagulation |
|||
K10/RCTPPC |
1 / vitesse d’organisation du gel en PPC |
0,62 |
0,06 |
aPPC |
Fermeté du gel après une fois le temps de prise en PPC |
0,72 |
1,4 |
K10/RCTPM |
1 / vitesse d’organisation du gel en PM |
0,76 |
0,07 |
aPM |
Fermeté du gel après une fois le temps de prise en PM |
0,80 |
1,5 |
a2PM |
Fermeté du gel après 2 fois le temps de prise en PM |
0,76 |
1,4 |
Acidification |
|||
pH0 PPC |
pH lait après ensemencement en PPC |
0,68 |
0,04 |
a PPC : Pâte Pressée Cuite, PM : Pâte Molle, RCT : « Rennet Coagulation Time » i.e. temps de coagulation ; b précision de l’équation de prédiction (n = 291) évaluée par le carré de la corrélation entre les valeurs vraies et les valeurs prédites (R²VAL) et l’écart-type d’erreur (SEVAL) dans une population de validation (n = 125).
1.3. Base de données spectrales
Le jeu de données initial, résultant du stockage systématique des spectres par trois entreprises de conseil en élevage (Doubs et Territoire de Belfort, Jura et Haute-Saône) depuis plusieurs années, comprenait 6 670 769 spectres MIR de 410 622 vaches Montbéliarde de la zone AOP Comté. Les échantillons de lait ont été prélevés dans le cadre du contrôle laitier entre janvier 2012 et juin 2017 et analysés par 10 spectromètres différents de type MilkoScan FT6000 (Foss Electric A/S, Hillerød, Danemark). Les spectres MIR produits ont été standardisés selon la procédure issue du projet OptiMIR (Grelet et al., 2016). Les équations de prédiction de la « fromageabilité » développées dans le projet From’MIR, ainsi que celles développées dans d’autres projets pour la composition du lait (protéines, projet PhénoFinlait ; acides gras et minéraux, projet OptiMIR) ont été appliquées sur les données spectrales. Les contrôles avant 7 jours et au-delà de 350 jours de lactation et les lactations incomplètes ont été éliminés. Les prédictions extrêmes hors de l’intervalle moyenne ± 4 écart-types ont également été éliminées. In fine, un peu plus de 4,8 millions de prédictions de 311 613 vaches provenant de 3 229 élevages franc-comtois (1 753 du Doubs et du Territoire de Belfort, 865 du Jura et 611 de la Haute-Saône) ont ainsi été conservées.
2. Paramètres génétiques des aptitudes fromagères du lait
Les paramètres génétiques ont été estimés en considérant différents jeux de données et différents modèles pour répondre à un certain nombre de questions concernant le déterminisme génétique des aptitudes fromagères du lait.
2.1. Dans quelle mesure les aptitudes fromagères sont-elles héritables et quels sont leurs liens génétiques avec la composition du lait ?
Pour répondre à cette question, nous avons étudié les caractères à l’échelle du contrôle en première lactation (1,1 millions de prédictions MIR de 126 873 vaches) et avons estimé les héritabilités des aptitudes fromagères et les corrélations génétiques des aptitudes fromagères entre elles et avec les caractères de composition du lait .
Les estimations des héritabilités des neuf caractères fromagers et des corrélations génétiques entre ces caractères sont présentées dans le tableau 2. Les héritabilités des rendements fromagers et du pH sont modérées (0,37 à 0,39), celles des paramètres de coagulation sont un peu plus fortes (0,42 à 0,48). Ces valeurs sont globalement élevées pour des caractères mesurés par contrôle. Les caractères moyens par lactation ont des héritabilités encore plus élevées (0,70-0,73 en première lactation). Par ailleurs, les coefficients de variation génétique (rapport entre l’écart-type génétique et la moyenne du caractère), indicateurs de l’étendue de la variabilité génétique du caractère, sont très variables selon le caractère : entre 0,5 % pour pH0 PPC et 13,8 % pour K10/RCTPM.
Tableau 2. Héritabilités (diagonale en italique), corrélations génétiques (hors diagonale) et coefficients de variation génétique (CVGEN) des aptitudes fromagères.
|
RDT_FRAIS |
RDT_ES |
RDT_MSU |
K10/RCTPPC |
aPPC |
K10/RCTPM |
aPM |
a2PM |
pH0 PPC |
|---|---|---|---|---|---|---|---|---|---|
RDT_FRAIS * |
0,38 |
0,97 |
– 0,84 |
– 0,73 |
0,78 |
– 0,76 |
0,78 |
0,75 |
0,03 |
RDT_ES |
|
0,39 |
– 0,82 |
– 0,72 |
0,77 |
– 0,74 |
0,76 |
0,72 |
– 0,02 |
RDT_MSU |
|
0,37 |
0,65 |
– 0,65 |
0,71 |
– 0,67 |
– 0,64 |
– 0,01 |
|
K10/RCTPPC |
|
0,42 |
– 0,76 |
0,80 |
– 0,73 |
– 0,72 |
0,09 |
||
aPPC |
|
0,47 |
– 0,78 |
0,76 |
0,77 |
– 0,08 |
|||
K10/RCTPM |
|
0,45 |
– 0,77 |
– 0,77 |
0,06 |
||||
aPM |
|
0,48 |
0,74 |
0,02 |
|||||
a2PM |
|
0,47 |
0,02 |
||||||
pH0 PPC |
|
|
|
|
|
|
|
|
0,37 |
CVGEN (%) |
8,2 |
3,3 |
4,4 |
12,0 |
6,3 |
13,8 |
6,6 |
4,3 |
0,5 |
* RDT_FRAIS : rendement frais ; RDT_ES : rendement en extrait sec ; RDT_MSU : rendement en matière sèche utile ; K10/RCTPPC : 1 / vitesse d’organisation du gel en Pâte Pressée Cuite (PPC) ; aPPC : fermeté du gel après une fois le temps de prise en PPC ; K10/RCTPM : 1 / vitesse d’organisation du gel en Pâte Molle (PM) ; aPM : fermeté du gel après une fois le temps de prise en PM ; a2PM : fermeté du gel après 2 fois le temps de prise en PM ; pH0 PPC : pH lait après ensemencement en PPC.
Concernant les corrélations génétiques, il est important de noter tout d’abord qu’elles sont toutes favorables, qu’elles soient positives ou négatives. En effet, le signe dépend de la définition des caractères. Ainsi, pour obtenir un lait plus « fromageable », on cherche à diminuer le rendement en matière sèche utile (RDT_MSU, exprimé comme la quantité de MSU du lait nécessaire pour obtenir une quantité d’extrait sec de caillé) et à augmenter la vitesse de coagulation, et donc à diminuer K10/RCT. Dans le texte qui suit, on s’intéressera donc aux valeurs absolues des corrélations génétiques. Globalement, elles sont très fortes entre les rendements fromagers (0,82 à 0,97), entre les paramètres de coagulation (0,72 à 0,80) et dans une moindre mesure entre les rendements fromagers et les paramètres de coagulation (0,64 à 0,78). On note également de fortes corrélations génétiques entre les paramètres de coagulation mesurés en PPC et en PM (0,72 à 0,80). En revanche, le seul paramètre d’acidification prédit par la spectrométrie MIR (pH0 PPC) semble génétiquement indépendant des autres caractères fromagers.
Les rendements fromagers et les paramètres de coagulation sont également étroitement liés à la composition du lait, exprimée en pourcentage de lait (tableau 3). De plus fortes proportions de protéines, acides gras et minéraux dans le lait sont associées à un lait présentant de meilleures aptitudes fromagères. De même que précédemment, nous nous focaliserons sur les valeurs absolues des corrélations. Les rendements frais et en extrait sec sont génétiquement très fortement corrélés au TB (0,87 et 0,91 respectivement) et aux composants fortement corrélés au TB comme les acides gras saturés. Ces mêmes rendements sont génétiquement un peu moins fortement corrélés au TP (0,75 et 0,74 respectivement) et aux caséines qui constituent environ 80 % des protéines du lait. Pour les paramètres de coagulation, les corrélations génétiques sont beaucoup plus fortes avec le TP ou les caséines (0,80 à 0,94) qu’avec le TB (0,47 à 0,55). En revanche, contrairement aux rendements, les paramètres de coagulation sont génétiquement plus fortement corrélés aux acides gras insaturés qu’aux saturés. Les teneurs en calcium et phosphore du lait sont modérément corrélées avec les rendements frais et en extrait sec (0,25 à 0,58) et avec les paramètres de coagulation (0,37 à 0,59).
Tableau 3. Corrélations génétiques entre les aptitudes fromagères et la composition du lait exprimée en % de lait.
|
Protéines |
Acides gras (AG) |
Minéraux |
|||||
|---|---|---|---|---|---|---|---|---|
Totales |
Caséines |
Protéines |
Totaux |
AG |
AG |
Calcium |
Phosphore |
|
RDT_FRAIS * |
0,75 |
0,75 |
0,26 |
0,87 |
0,81 |
0,72 |
0,40 |
0,58 |
RDT_ES |
0,74 |
0,74 |
0,23 |
0,91 |
0,86 |
0,71 |
0,41 |
0,54 |
RDT_MSU |
– 0,52 |
– 0,54 |
0,09 |
– 0,57 |
– 0,52 |
– 0,52 |
– 0,25 |
– 0,44 |
K10/RCTPPC |
– 0,80 |
– 0,80 |
– 0,45 |
– 0,53 |
– 0,47 |
– 0,53 |
– 0,46 |
– 0,58 |
aPPC |
0,94 |
0,94 |
0,41 |
0,55 |
0,49 |
0,53 |
0,45 |
0,59 |
K10/RCTPM |
– 0,81 |
– 0,81 |
– 0,40 |
– 0,47 |
– 0,40 |
– 0,52 |
– 0,37 |
– 0,58 |
aPM |
0,91 |
0,90 |
0,38 |
0,51 |
0,45 |
0,52 |
0,39 |
0,54 |
a2PM |
0,89 |
0,90 |
0,38 |
0,48 |
0,42 |
0,50 |
0,42 |
0,54 |
pH0 PPC |
– 0,11 |
– 0,12 |
– 0,05 |
– 0,06 |
– 0,08 |
0,00 |
0,10 |
– 0,18 |
* Voir la définition des caractères dans la légende du tableau 2 ; en gras : valeur absolue de la corrélation > 0,80 ; en italique : valeur absolue de la corrélation < 0,20.
Ces résultats sont cohérents avec ceux obtenus précédemment à partir de mesures de référence (Bittante et al., 2013) ou de prédictions MIR (Bonfatti et al., 2017) dans d’autres races.
2.2. Le déterminisme génétique des aptitudes fromagères est-il le même tout au long de la lactation et entre lactations ?
Les résultats présentés précédemment supposent que le caractère est génétiquement le même tout au long de la lactation. Nous avons vérifié cette hypothèse en appliquant un modèle de régression aléatoire sur le même jeu de données (Schaeffer, 2004). Pour chaque caractère, un tel modèle permet d’estimer la trajectoire d’héritabilité au cours de la lactation ainsi que les corrélations entre stades de la lactation. Les trajectoires obtenues pour le rendement en extrait sec sont représentées dans la figure 1. L’héritabilité varie au cours de la lactation, elle est minimale au tout début (0,25), maximale vers 150 jours (0,52) puis elle diminue légèrement jusqu’en fin de lactation (0,42). Les corrélations génétiques entre le RDT_ES mesuré d’une part à 5 stades de lactation différents (7, 92, 178, 264 et 350 jours) et d’autre part chaque jour de lactation compris entre 7 et 350, sont positives et comprises entre 0,55 et 1. Les corrélations sont toujours très élevées entre stades proches et pour la majorité des combinaisons. Elles sont en revanche plus faibles entre le début (7 j) et le reste de la lactation et, dans une moindre mesure, entre la fin (350 j) et le reste de la lactation.
Figure 1. Courbe phénotypique moyenne et trajectoire d’héritabilités (à gauche) et trajectoires de corrélations génétiques entre jours de lactation (à droite) pour le rendement en extrait sec entre 7 et 350 jours de lactation.

On observe les mêmes évolutions de paramètres génétiques au cours de la lactation pour les autres caractères fromagers. La matrice de variance – covariance de la trajectoire de chacun des caractères présente une première valeur propre qui explique en moyenne 82 % de leur variabilité génétique. Le vecteur propre correspondant qui combine chaque stade avec des coefficients positifs et peu différents reflète donc le niveau moyen du caractère au cours de la lactation. Ce résultat montre que les caractères fromagers sont génétiquement homogènes au cours de la lactation. L’utilisation d’un modèle plus simple, à l’échelle du contrôle ou de la lactation, est donc justifiée. Le deuxième vecteur propre reflète les écarts entre début et fin de lactation et s’interprète comme une persistance.
Après avoir montré que le déterminisme génétique des aptitudes fromagères était assez homogène au cours de la première lactation, nous l’avons étudié sur les trois premières lactations (L1, L2 et L3). Pour chaque caractère fromager, le phénotype a été défini par la moyenne des performances par lactation pour chaque vache. Les moyennes en L1 (145 861 vaches), L2 (77 182 vaches) et L3 (36 957 vaches) ont été considérées comme trois caractères différents dont nous avons estimé les corrélations génétiques. Elles sont très fortes pour tous les caractères fromagers : en moyenne 0,97 entre la L1 et la L2, 0,96 entre la L1 et la L3 et 0,99 entre la L2 et la L3. Ces résultats montrent que les gènes impliqués dans le déterminisme des aptitudes fromagères sont les mêmes en première, deuxième et troisième lactation et valident donc l’utilisation des performances sur une lactation pour des études génétiques.
2.3. Les aptitudes fromagères du lait présentent-elles des antagonismes génétiques avec les caractères actuellement sélectionnés en race Montbéliarde ?
Les caractères fromagers présentent des valeurs d’héritabilité et de coefficients de variation génétique tout à fait comparables à celles estimées pour les taux protéique et butyreux par exemple qui sont sélectionnés avec efficacité depuis de nombreuses années. Il devrait donc être possible de sélectionner assez facilement les aptitudes fromagères du lait. Toutefois, il faut auparavant s’assurer qu’elles ne présentent pas de corrélations génétiques défavorables avec les autres caractères qui composent l’objectif de sélection en race Montbéliarde. Pour cela, en considérant les performances moyennes en première lactation, nous avons estimé les corrélations génétiques entre d’une part, les aptitudes fromagères et d’autre part, la production laitière (quantités de lait, de matières grasse et de matière protéique), la santé de la mamelle (score de cellules somatiques), le taux de réussite à l’insémination post-partum, le développement de la mamelle (apprécié lors du pointage morphologique) et la vitesse de traite. Hormis une corrélation génétique défavorable modérée avec la production laitière, qui varie entre 0,32 et 0,47 selon le caractère fromager, aucun antagonisme fort génétique n’est observé avec les autres caractères (valeurs absolues comprises entre 0,01 et 0,12).
3. Gènes et variants affectant les aptitudes fromagères du lait
3.1. Génotypages et imputations des génotypes manquants
Grâce aux puces à ADN, disponibles depuis 2008 dans l’espèce bovine, il est possible d’obtenir les génotypes pour de nombreux SNP (« Single Nucleotide Polymorphism » = variation d’un seul nucléotide sur la séquence de l’ADN) répartis sur tout le génome à partir d’une analyse effectuée sur un échantillon de sang ou de cartilage prélevé sur l’animal. Il existe des puces de différentes densités, permettant notamment de génotyper 10 000 (EuroG10K), 50 000 (50K) ou 700 000 (HD) SNP. En mettant en évidence des associations entre régions du génome et caractères, les génotypes aux SNP sont des outils de choix pour identifier les gènes et les variations dans ces gènes responsables des effets sur les caractères (gènes et variants causaux). Le projet From’MIR a pu bénéficier des génotypages de 19 586 vaches effectués pour la sélection génomique par les éleveurs adhérents à l’entreprise Umotest (6 505 avec la puce 50K et 13 081 avec la puce EuroG10K). À noter que la puce EuroG10K, développée par et pour le consortium Eurogenomics (8 pays européens dont la France) contient également une partie dite « recherche » de plusieurs milliers de variants candidats en cours d’étude, en plus des 10 000 SNP génériques.
Même si leur densité en SNP peut être élevée, les puces commerciales disponibles ne donnent pas accès à l’intégralité des variations du génome et donc pas à tous les variants causaux. Une solution est d’imputer les variants absents de la puce par une méthode statistique utilisant l’information familiale ainsi que celle du déséquilibre de liaison entre variants. Plus les animaux de la population de référence sont nombreux et proches des animaux à imputer (de même race par exemple), meilleure est la précision de l’imputation. C’est cette approche qui a été utilisée, en s’appuyant sur les données de séquence du génome de 2 333 bovins, obtenus dans le cadre du projet international « 1 000 génomes bovins » (Daetwyler et al., 2014). La liste des variants (SNP ou insertions-délétions de quelques nucléotides) étant proche de l’exhaustivité, elle contient en théorie les variants causaux qui ont des effets sur les caractères, ce qui permet de les cibler directement dans les analyses d’association.
3.2. Inventaire et fréquence des variants des lactoprotéines à partir des génotypages
Les variants des principales protéines du lait de vache ont très tôt suscité l’intérêt en raison de leurs effets forts sur la production, la composition et la « fromageabilité » du lait (Grosclaude, 1988). On connaît aujourd’hui les gènes et les variations dans ces gènes qui induisent les variants des lactoprotéines. Les gènes des caséines sont groupés dans une région de 250 kb sur le chromosome 6 (entre 87,1 et 87,4 Mb). On trouve dans l’ordre, les gènes des caséines s1 (CSN1S1), (CSN2), s2 (CSN1S2) et (CSN3). Les gènes qui codent pour les protéines sériques -lactalbumine et -lactoglobuline sont respectivement situés sur les chromosomes 5 (LALBA) à 31,3 Mb et 11 (PAEP) à 103,3 Mb. Peu de variants existent dans les gènes LALBA et CSN1S2 dans les races bovines et en particulier dans les races laitières françaises alors que les quatre autres gènes (CSN1S1, CSN2, CSN3 et PAEP) présentent de nombreux polymorphismes. En raison de leurs effets avérés ou potentiels sur les caractères laitiers, plusieurs SNP localisés dans les gènes CSN1S1, CSN2, CSN3 et PAEP sont présents sur la puce EuroG10K (Boichard et al., 2018). Treize de ces SNP présents sur la puce sont non synonymes et induisent un changement d’un acide aminé de la protéine. Ils permettent de caractériser les principaux variants protéiques (2 à 5) de chaque gène dans les populations actuelles, en particulier en race Montbéliarde pour laquelle les génotypes de ces SNP sont disponibles pour 132 387 animaux (dont les 19 586 vaches From’MIR génotypées). Il a par ailleurs été possible d’imputer les génotypes de ces SNP pour 3 224 taureaux de race Montbéliarde nés entre 1996 et 2015 et de suivre l’évolution de la fréquence des variants par année de naissance des taureaux (figure 2).
Dans le gène CSN1S1, deux variants seulement sont caractérisables et le variant B est largement majoritaire (97,5 %). Dans le gène CSN2, cinq variants sont caractérisés, mais avec les génotypages disponibles, il n’a pas été possible de distinguer les variants A1, C et F. Quatre variants sont majoritaires : le plus fréquent est le variant A2 (51,6 %), puis le variant A1 (+C et F) (26,6 %), le variant B (11,1 %) et le variant I (10,7 %). Dans le gène CSN3, deux variants sont majoritaires : A et B, le variant B étant le plus fréquent (61,3 %). Enfin, dans le gène PAEP, essentiellement deux variants A et B coexistent avec des fréquences relativement équilibrées, respectivement 54,4 et 45,5 %. L’évolution des fréquences, calculées par année de naissance des taureaux (1996-2015), permet d’évaluer l’impact de la sélection au cours du temps. Globalement, les fréquences des variants de la caséine s1 varient peu au fil des ans. Pour la caséine , la fréquence du variant I, très faible dans les années 1990, augmente régulièrement jusqu’à aujourd’hui pour atteindre 17 %. Pour la caséine , la fréquence du variant B, plus faible au départ, augmente régulièrement au détriment de celle du variant A. On observe sensiblement la même tendance pour les fréquences des variants A et B de la -lactoglobuline : le variant A, légèrement minoritaire dans les années 1990, est aujourd’hui le plus fréquent.
Figure 2. Principaux variants des lactoprotéines en race Montbéliarde : a) fréquences dans la population actuelle et b) évolution des fréquences pour les taureaux nés entre 1996 et 2015.

Si les fréquences calculées sur les taureaux nés en 1996 sont tout à fait cohérentes avec celles estimées à la fin des années 1970 , nous montrons un état des lieux actuel sensiblement différent avec notamment l’émergence de deux nouveaux variants qui semblent avoir été sélectionnés indirectement depuis le milieu des années 2000 : le variant C de la caséine s1 et le variant I de la caséine . Cette étude met également en évidence l’augmentation de la fréquence du variant A de la -lactoglobuline qui pourrait avoir des effets défavorables sur la fromageabilité du lait et notamment sur les paramètres de coagulation (Caroli et al., 2009).
3.3. Recherche des régions du génome (QTL) affectant les caractères
La population séquencée dans le run6 du projet « 1 000 génomes bovins », contient 2 333 animaux, dont les 54 ancêtres majeurs de la race Montbéliarde. À partir de cette population, les séquences complètes des 19 586 vaches du projet From’MIR ont été reconstituées. Pour cela, l’imputation a été réalisée en plusieurs étapes de la puce EuroG10K vers la puce 50 K, puis de la puce 50 K vers la puce HD et enfin de la puce HD jusqu’à la séquence. Les génotypes de 27 millions de variants ont ainsi été imputés. Après élimination des variants avec les précisions d’imputation les plus faibles (R² < 0,20) ou peu polymorphes (fréquence de l’allèle le plus rare ou MAF pour « Minor Allele Frequency » <1 %), 8 551 748 variants ont été analysés.
Les caractères de « fromageabilité » et de composition du lait mesurés sur les vaches avec au moins 3 contrôles en première lactation (1 442 371 contrôles de 189 817 vaches) ont été utilisés. Ces performances ont été corrigées pour les effets non génétiques du groupe de contemporaines (effet combiné du troupeau, du jour de contrôle et du spectromètre), de l’âge au vêlage et du stade de lactation estimés dans un modèle incluant aussi les effets aléatoires génétique et de l’environnement permanent de l’animal. Pour chaque caractère, le phénotype de chaque vache a été défini comme la moyenne de ses performances corrigées des effets non génétiques.
Avec une population d’animaux à la fois génotypés et phénotypés (i.e. avec performances), il est possible de rechercher les régions du génome, ou QTL pour « Quantitative Trait Loci », qui ont des effets sur les caractères d’intérêt. L’approche la plus communément employée est l’analyse d’association (GWAS pour « Genome Wide Association Study »), implémentée dans de nombreux logiciels dont GCTA (Yang et al., 2011) utilisé ici. L’effet fixé de chaque variant est estimé dans un modèle incluant également l’effet aléatoire génétique de l’animal, estimé via une matrice de parenté génomique calculée à partir des SNP de la puce HD, ce qui permet d’éviter des biais éventuels liés à la structure de la population. La statistique utilisée pour chaque variant est un test de Student. Le nombre de faux positifs lié au grand nombre de tests est contrôlé par une correction de Bonferroni. Cette méthode est très sévère puisqu’elle considère les tests indépendants. Un test est ainsi considéré significatif si P < 5,8.10-9 ou –log10(P) > 8,2.
Nous avons ainsi détecté 59 régions QTL avec des effets significatifs sur les caractères fromagers ou de composition du lait. Ces régions QTL de taille variable contiennent 6 à 401 variants avec des effets significatifs et sont réparties sur tous les autosomes du génome bovin, excepté les chromosomes 8 et 23. Presque toutes les régions QTL (56/59) contiennent un ou plusieurs gènes candidats. Environ 60 % (4 312/7 393) des variants des régions QTL sont localisés dans ou à proximité (régions en amont ou en aval) de 264 gènes. Seuls 51 de ces variants sont non synonymes et sont donc responsables d’un changement d’acide aminé de la protéine. La majorité des variants (2 972) est localisée dans les introns des gènes.
Les valeurs des –log10(P) en fonction des positions des variants sur le génome sont représentées dans la figure 3 pour le rendement en extrait sec et un paramètre de coagulation. De tels graphiques, appelés aussi Manhattan Plot, nous permettent de visualiser les variants avec des effets significatifs (en vert dans la figure 3) qui forment des pics. En effet, en raison du DL entre les marqueurs proches, les tests statistiques sont corrélés entre eux et plusieurs variants avec des effets significatifs co-localisent généralement dans une même région QTL. Parmi eux, le variant causal devrait être en espérance le plus significatif et donc se trouver en haut du pic mais ce n’est pas toujours le cas, en raison notamment de l’imprécision des imputations des génotypes. Dans les régions QTL avec les effets les plus significatifs, des gènes candidats sont mis en évidence sur les chromosomes 1 (SLC37A1), 2 (ALPL), 4 (CBLL1), 5 (MGST1), 6 (SEL1L3 et gènes des caséines), 7 (FSTL4), 11 (PAEP), 12 (ABCC4), 14 (DGAT1 et GPT), 17 (BRI3BP), 19 (FASN), 20 (ANKH), 22 (FAM19A4 et KLF15), 25 (FAM57B), 26 (SCD), 27 (GPAT4) et 29 (EED). La puissance du dispositif et le large panel de caractères analysés dans le projet From’MIR permettent de confirmer l’effet de certains gènes identifiés dans le projet PhénoFinlait pour la composition en acides gras ou en protéines (Boichard et al., 2014 ; Sanchez et al., 2017) et de mettre en évidence de nouveaux gènes affectant les caractères de « fromageabilité » et de composition du lait.
Figure 3. Résultats des détections de QTL pour le rendement en extrait sec (RDT_ES) et un paramètre de coagulation (aPM) : -log10(P) en fonction de la position des variants sur les 29 autosomes du génome bovin.
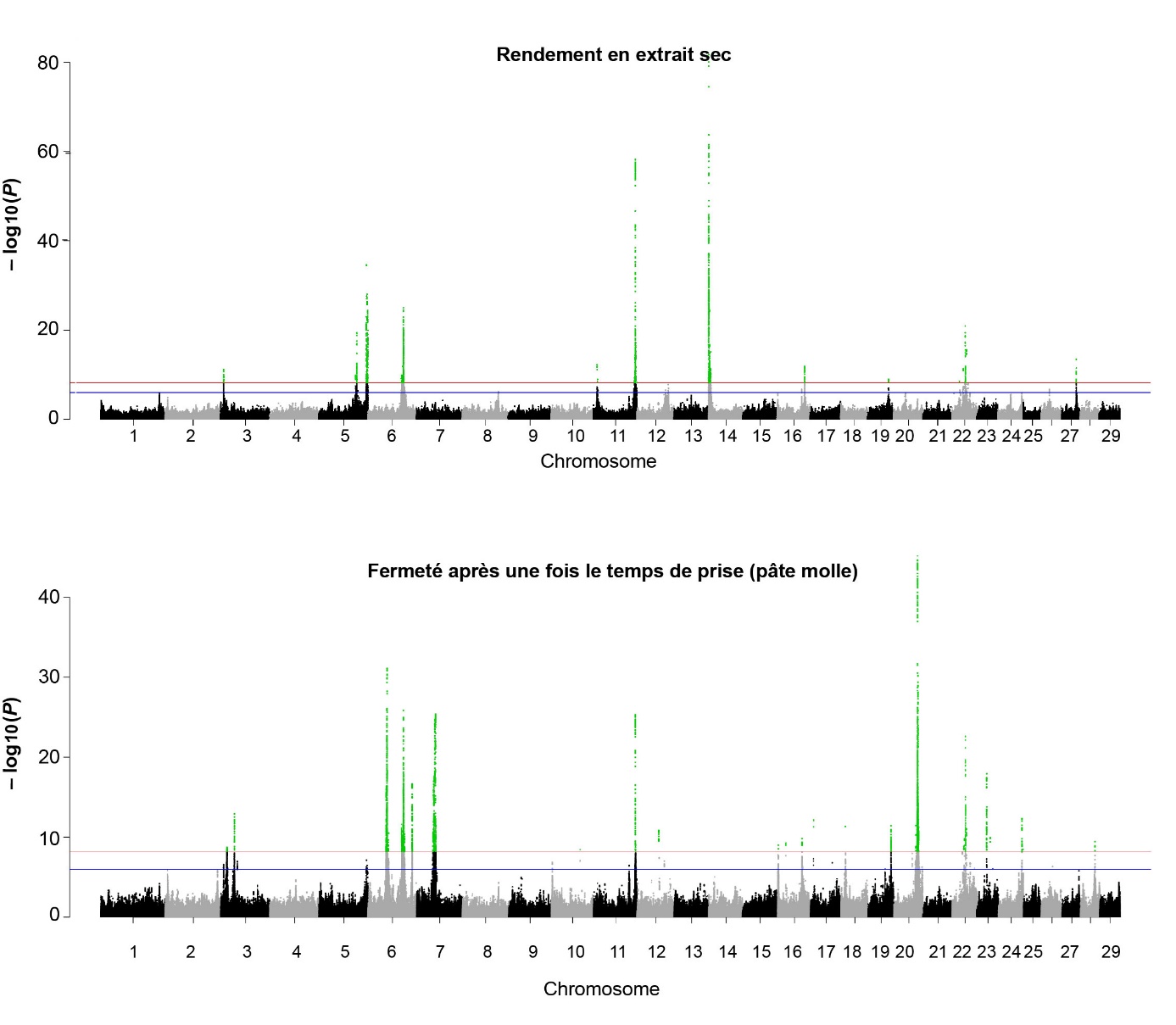
Selon le caractère analysé, le nombre de QTL détectés (6 à 19) et le pourcentage de la variance phénotypique expliquée par ces QTL (4,7 à 62,4 %) sont très variables. Le caractère le plus polygénique est le paramètre de coagulation aPM (figure 3) pour lequel nous détectons 19 QTL expliquant chacun entre 0,2 et 1,9 % de la variance phénotypique. Au contraire, nous mettons en évidence seulement 6 QTL pour le taux de protéines sériques. Pour ce caractère, le QTL localisé dans la région du gène PAEP présente les effets les plus forts et explique à lui seul 56 % de la variance phénotypique du taux de protéines sériques.
3.4. Analyse de réseaux de gènes
À partir des résultats de GWAS, nous avons appliqué l’approche AWM-PCIT (« Association Weight Matrix » (Fortes et al., 2010) – « Partial Correlation and Information Theory » (Reverter et Chan, 2008)) pour mettre en évidence un réseau de gènes coassociés aux caractères étudiés. Le principe de l’AWM est de sélectionner parmi l’ensemble des résultats des analyses GWAS (effets et probabilités associées à tous les variants pour tous les caractères) un jeu de variants associés à des gènes et représentatif de la variabilité génétique des caractères. Pour cela, les étapes décrites ci-après ont successivement été réalisées sur les données From’MIR :
i) définition d’un phénotype clé => rendement en extrait sec ;
ii) sélection des variants avec des effets significatifs avec un seuil peu sévère (P < 0,001) sur le phénotype clé => 38 858 variants ;
iii) calcul du nombre moyen de caractères présentant des effets significatifs pour les 38 858 variants sélectionnés en 2) => 6 caractères ;
iv) sélection des variants avec des effets significatifs sur le phénotype clé ou sur au moins 6 caractères corrélés (corrélation ≥ 0,25) au phénotype clé => 41 180 variants (38 858 + 2 322) ;
v) sélection des variants associés à des gènes (distance maximale = 10 kb) => 15 330 variants ;
vi) sélection d’un variant par gène (celui associé au plus grand nombre de caractères ou si égalité, celui qui présente la somme des probabilités la plus faible) => 736 variants.
On obtient ainsi un réseau de 736 gènes co-associés aux caractères de « fromageabilité » et de composition du lait. Les corrélations entre caractères calculées à partir des effets des 736 variants retenus sont très proches des corrélations génétiques estimées à partir du pedigree, ce qui suggère que le réseau de gènes est représentatif du déterminisme génétique des caractères et qu’il en explique sans doute une grande partie. L’approche PCIT qui calcule les corrélations partielles pour chaque trio de gènes, met en évidence 59 168 interactions significatives entre les 736 gènes.
Le réseau est ensuite étudié via différents outils. Le logiciel Cytoscape (Shannon et al., 2003) et ses différents « plug-in », notamment iRegulon (Janky et al., 2014) et ClueGO (Bindea et al., 2009), met en évidence les régulateurs (facteurs de transcription, micro-ARN…) et les voies métaboliques associés à ce réseau de gènes. Le facteur de transcription ASXL3 qui régule la lipogenèse et le micro-ARN bta-mir-200c, hautement exprimé dans la glande mammaire et présent dans le lactosérum, sont parmi les régulateurs qui interagissent avec le plus grand nombre de gènes du réseau (276 et 240 gènes respectivement). Nous montrons également que des processus biologiques liés notamment au transport du potassium, au métabolisme du phosphate et des phospholipides et à la voie de signalisation du calcium sont enrichies en gènes du réseau. Les gènes SLC37A1 et ANKH, meilleurs candidats pour les QTL identifiés par GWAS sur les chromosomes 1 et 20 et qui codent respectivement un transporteur du glucose-6-phosphate et une protéine transmembranaire impliquée dans le transport des ions, sont trouvés co-régulés et co-associés via cette dernière approche.
Le réseau de gènes identifié par l’approche AWM-PCIT a une signification biologique en lien avec les caractères étudiés dans le projet From’MIR (Sanchez et al., 2019).
4. Évaluation génomique des aptitudes fromagères du lait
Les caractères de « fromageabilité » et de composition du lait étant héritables et sous l’influence de gènes avec des effets parfois très forts, ils doivent pouvoir être sélectionnés assez facilement. Par ailleurs, avec environ 20 000 vaches Montbéliardes à la fois phénotypées et génotypées, le projet From’MIR offre une première population de référence de taille conséquente pour envisager une sélection génomique. Dans cette partie, pour 11 caractères (les 9 caractères fromagers les mieux prédits par la spectrométrie MIR et les taux de caséines (CN) et de calcium (Ca) dans le lait), nous avons étudié i) la précision d’une évaluation génomique, ii) le progrès génétique réalisé ces 13 dernières années et iii) les potentialités de sélection.
4.1. Précision d’une évaluation génomique
Compte tenu du jeu de données From’MIR (un grand nombre de vaches avec phénotypes, dont une partie génotypée), la précision d’une évaluation génomique a été évaluée sur une population de validation à partir d’une méthode en une étape (SS-GBLUP pour « Single Step – Genomic Best Linear Unbiased Prediction »). Cette méthode combine l’information apportée par tous les animaux, qu’ils soient génotypés ou non.
a. Populations d’apprentissage et de validation
À partir des données From’MIR décrites au 1.3, nous avons sélectionné les vaches avec un âge au premier vêlage compris entre 22 et 44 mois, une première lactation complète et au moins trois contrôles. Seuls les contrôles entre 7 et 305 jours de lactation ont été retenus et les prédictions au-delà de 3 écart-types autour de la moyenne ont été éliminées. Le jeu de données final comprenait 2 869 353 contrôles de 191 532 vaches (toutes lactations) parmi lesquelles 19 564 étaient génotypées. Le pedigree, constitué en remontant quatre générations, contenait 423 348 animaux.
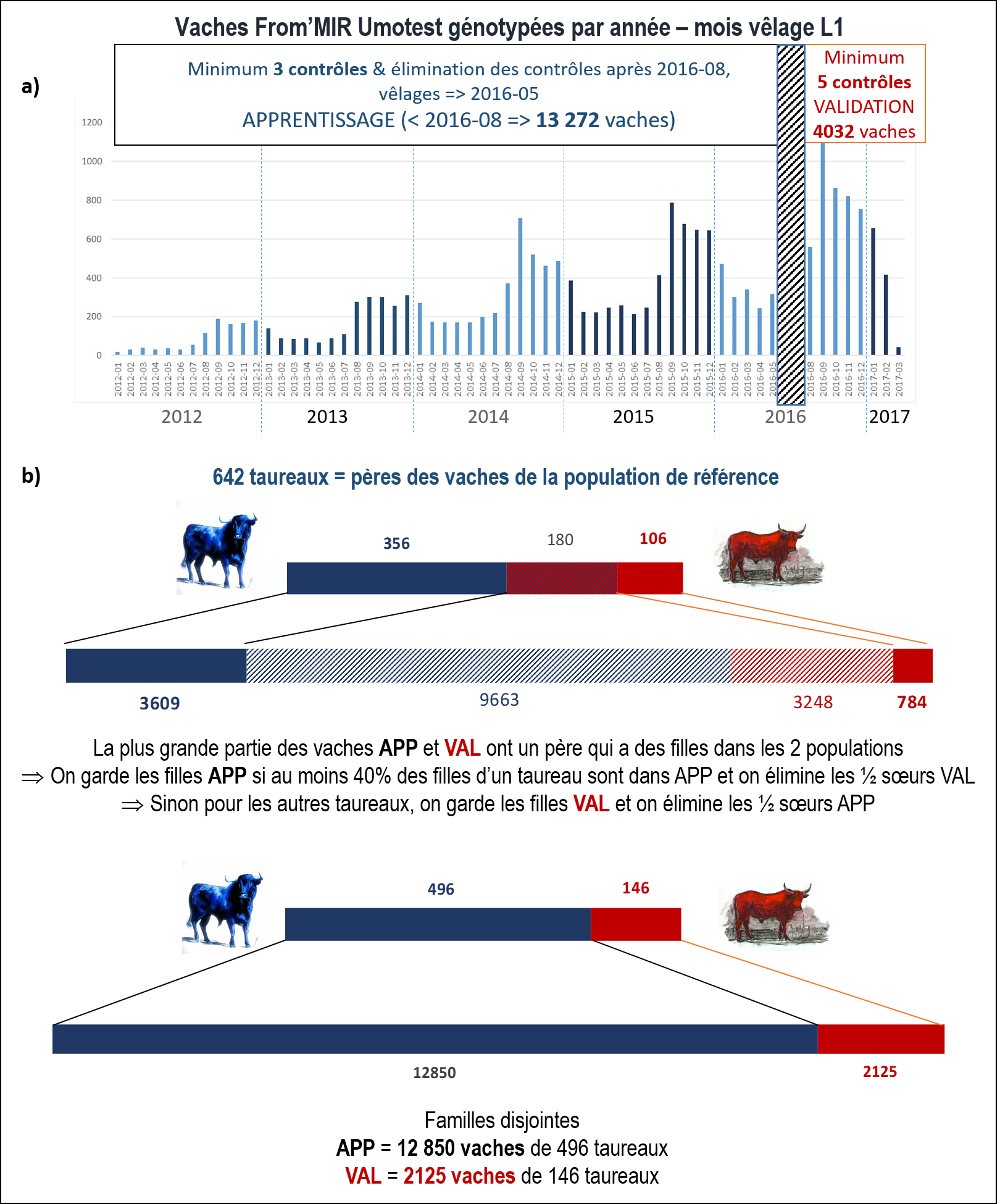
La précision de l’évaluation a été estimée sur une population de validation, en comparant l’évaluation génomique de ces animaux à leurs performances non prises en compte dans l’évaluation. À partir des 19 564 vaches avec phénotypes et génotypes, nous avons constitué une population d’apprentissage (APP) et une population de validation (VAL), plus récente et aussi indépendante que possible de la population d’apprentissage (figure 4). Les vaches primipares lors de la dernière campagne de vêlage (à partir d’août 2016) ont servi à constituer la population VAL, leurs performances ont donc été exclues des données utilisées pour l’évaluation génomique, ainsi que toutes les données contemporaines postérieures à août 2016. La population APP a par conséquent été définie avec les vaches primipares des campagnes précédentes (figure 4a). Les vaches des deux populations étaient les filles de 642 taureaux dont 180 avaient des filles à la fois dans la population APP et VAL. Pour éviter d’avoir des vaches très apparentées (demi-sœurs de même père) dans les populations APP et VAL, pouvant conduire à surestimer la précision de l’évaluation génomique, nous avons éliminé une partie de la descendance en ne conservant que la fraction APP ou VAL selon les familles. Les familles sélectionnées dans les deux populations étaient donc différentes (figure 4b). Les populations APP et VAL ont ainsi été créées avec des vaches ayant réalisé leur première lactation dans des campagnes différentes et issues de familles disjointes. In fine, la population APP se composait de 12 850 vaches génotypées et 147 069 vaches non génotypées, issues de 496 taureaux, et la population VAL contenait 2 125 vaches génotypées, filles de 146 autres taureaux.
Figure 4. Constitution des populations d’apprentissage (APP) et de validation (VAL) à partir des 19 564 vaches From’MIR Umotest phénotypées et génotypées, sélection des vaches sur a) les performances et b) les familles.
b. Méthode et modèles testés
La méthode d’évaluation génomique utilisée est la méthode dite HSSGBLUP (Hybrid-SS-GBLUP) décrite par Fernando et al. et implémentée à l’INRA. Elle a été appliquée à un modèle qui considère la performance moyenne de la vache sur la lactation (MOY équivalent au modèle utilisé au paragraphe 2.3) ou à un modèle à répétabilité qui prend en compte les performances de la vache à chacun de ses contrôles (CTL équivalent au modèle utilisé au paragraphe 2.1). Chaque type de modèle a été appliqué aux données des premières lactations (MOY1 et CTL1) ou aux données des trois premières lactations (MOY3 et CTL3). Quatre modèles différents ont ainsi été testés.
Grâce à l’imputation réalisée sur les vaches From’MIR (Sanchez et al., 2018b), nous avions à disposition les génotypes de la puce 50K augmentée des SNP de la partie recherche de la puce EuroG10K. Parmi les SNP de la partie recherche, figuraient certains variants candidats (prédictifs ou causaux) dont les effets sur la composition du lait ont été mis en évidence dans le projet PhénoFinlait (Boichard et al., 2014 ; Sanchez et al., 2017) et confirmés dans le projet From’MIR (Sanchez et al., 2018b). Dix-huit de ces variants (SNP), représentant chacun une région QTL avec de gros effets sur la composition et/ou la « fromageabilité » du lait, ont ainsi été sélectionnés. Selon le caractère, 5 à 14 de ces SNP présentaient des effets significatifs dans les GWAS.
Plusieurs jeux de marqueurs et pondérations associées à ces marqueurs ont donc été testés :
i) Les quatre modèles MOY1, MOY3, CTL1 et CTL3 ont été testés avec les SNP polymorphes de la puce 50K, soit 41 942 marqueurs. La variance des effets de ces SNP est supposée constante (Scénario 50K).
ii) Sur les quatre modèles, celui qui a conduit aux valeurs génomiques les plus précises a également été testé avec les 47 794 SNP polymorphes de la puce 50K et de la partie recherche de la puce EuroG10K avec des variances d’effets constantes pour chaque SNP (scénario 50K+) ou avec des variances spécifiques pour les effets de 5 à 14 SNP candidats sélectionnés selon le caractère.
Ces QTL expliquent de 17,5 à 58,4 % de la variance génétique du caractère. Les parts de variance génétique sont attribuées à chaque SNP à partir des effets estimés par les GWAS. Dans ce modèle, les autres SNP (47 780 à 47 789 selon le caractère) ont des variances d’effets constantes et se partagent le reste de la variance génétique, soit 41,6 à 82,5 % de la variance génétique totale selon le caractère (scénario QTL).
c. Estimation de la précision des valeurs génomiques
Le programme HSSGBLUP fournit une Valeur Génétique Estimée (VGE) pour chaque individu du pedigree. Les performances de la population de validation n’ayant pas été prises en compte pour estimer les VGE, la précision est estimée à partir de la corrélation entre les VGE et les performances corrigées pour les effets de milieu dans la population VAL en utilisant les modèles correspondants (MOY1, MOY3, CTL1 ou CTL3). Les coefficients de détermination (CD) sont calculés en divisant le carré de cette corrélation par l’héritabilité du caractère.
d. Comparaison de la précision des différents modèles
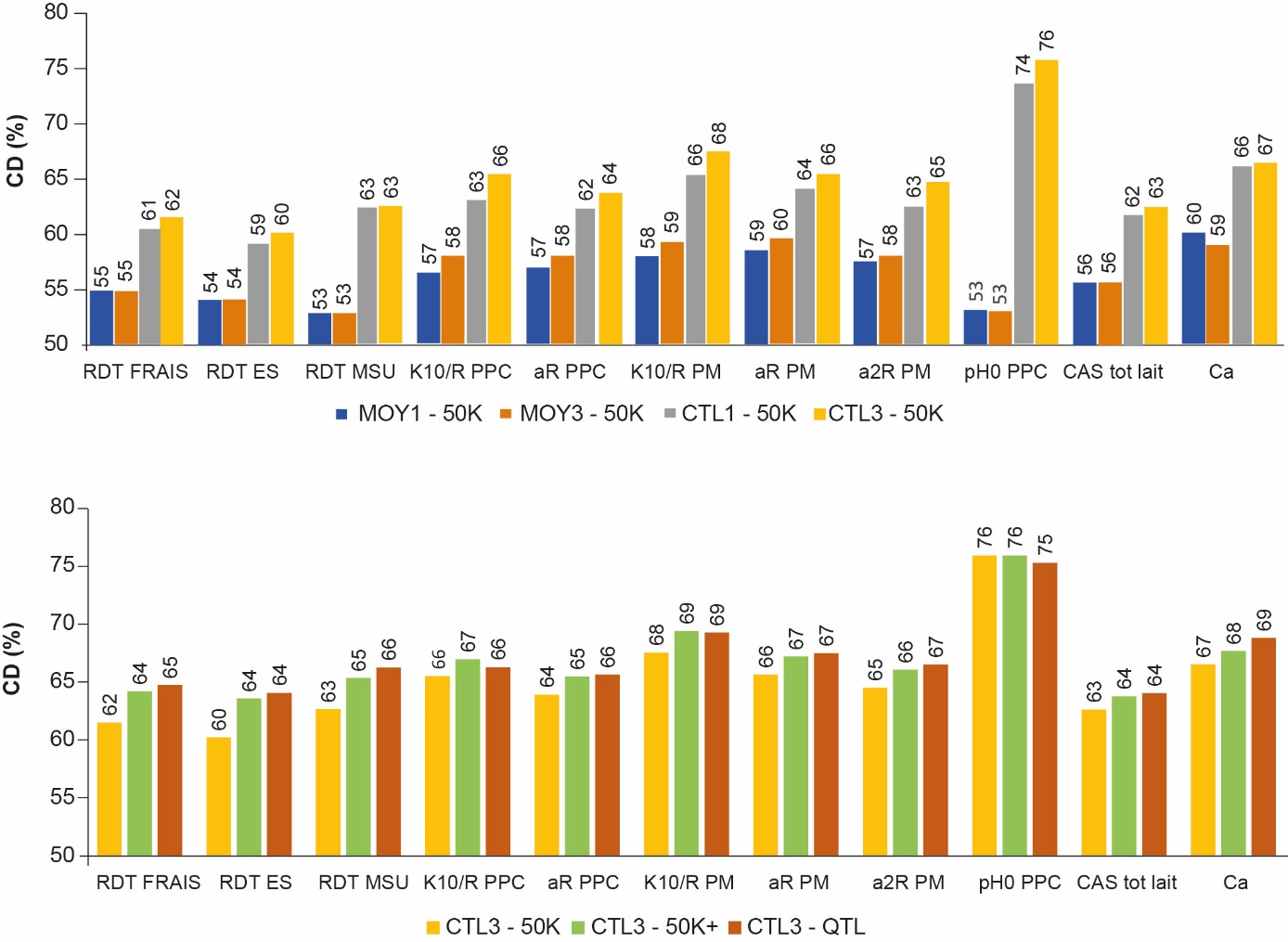
La figure 5a présente les valeurs de CD obtenues dans la population de validation pour les quatre modèles MOY1, MOY3, CTL1 et CTL3 (marqueurs de la puce 50K) et les 11 caractères analysés. Quel que soit le modèle testé et le caractère analysé, les CD sont toujours supérieurs à 50 %. Ce résultat est important en lui-même puisque le seuil de 50 % est le seuil de publication actuellement appliqué en race Montbéliarde pour les index génomiques des taureaux. Dans l’ensemble, les CD sont relativement proches pour tous les caractères. Pour un type de modèle donné (MOY ou CTL), la prise en compte des performances des deuxièmes et troisièmes lactations en plus de la première améliore légèrement la précision (+1 point de CD en moyenne). Dans tous les cas, les modèles de type contrôles élémentaires (CTL1 et CTL3) donnent des valeurs génomiques nettement plus précises que les modèles de type lactation (MOY1 et MOY3) : + 8 points de CD en moyenne. Sur les 11 caractères, les valeurs de CD moyennes sont égales à 56, 57, 64 et 65 % pour les modèles MOY1, MOY3, CTL1 et CTL3, respectivement.
Figure 5. Coefficients de détermination (CD en %) estimés dans la population de validation pour a) les 4 modèles et b) les 3 scénarios de prise en compte des SNP.
e. Comparaison de la précision des différents scénarios
Pour comparer les différents scénarios, nous avons sélectionné le modèle CTL3 qui donne les résultats les plus précis pour les 11 caractères (marqueurs 50K) et avons ré-estimé les valeurs génétiques en appliquant les scénarios 50K+ et QTL. La comparaison des CD pour les 11 caractères est présentée dans la figure 5b pour les trois scénarios 50K (mêmes valeurs qu’en figure 5a), 50K+ et QTL.
Bien que nous ayons déjà atteint de bons niveaux de CD avec les SNP de la puce 50K, l’ajout des marqueurs de la partie recherche de la puce EuroG10K (scénario 50K+ : 5852 SNP supplémentaires par rapport au scénario 50K) améliore encore la précision des valeurs génomiques. En moyenne sur tous les caractères, le gain est d’environ + 2 points de CD : + 2,7 à + 3,4 points pour les rendements fromagers et + 1,5 à + 1,9 points pour les paramètres de coagulation, les caséines et le calcium dans le lait. Le scénario QTL, qui attribue des pondérations plus élevées aux QTL, n’apporte qu’un gain supplémentaire de 0,25 point en moyenne, il permet par contre d’obtenir les VGE les moins biaisées (présentant des coefficients de régression des performances sur les VGE plus proches de 1).
En plus des variants candidats identifiés dans les projets PhénoFinlait et From’MIR, la partie recherche de la puce EuroG10K est particulièrement enrichie en SNP dans les régions QTL détectés pour les caractères de production laitière notamment (Boichard et al., 2018). Parmi ces régions QTL, on trouve par exemple les régions des gènes DGAT1, des caséines, PAEP, etc. qui ont également des effets forts sur les caractères fromagers. Cela explique probablement le gain de précision obtenu avec le scénario 50K+ par rapport au scénario 50K. On constate d’ailleurs que le gain est le plus fort pour les rendements fromagers qui sont génétiquement très corrélés au TB (Sanchez et al., 2018a) et donc particulièrement sous l’influence de la région du gène DGAT1.
Les modèles et scénarios comparés dans cette étude montrent que les valeurs génétiques les plus précises et les moins biaisées sont obtenues :
i) avec un modèle de type contrôles élémentaires appliqué aux trois premières lactations, qui utilise le maximum d’information et une modélisation plus précise du phénotype ;
ii) avec les génotypes aux marqueurs de la puce 50K et aux marqueurs de la partie recherche de la puce EuroG10K, option dans laquelle les marqueurs sont à la fois plus nombreux et relativement concentrés dans les régions QTL ;
iii) avec une pondération plus forte pour les variants causaux ou prédictifs.
Ces résultats sont obtenus avec une population de 12 000 vaches avec performances, les précisions seront donc encore plus élevées avec la population complète.
4.2. Progrès génétique réalisé sur les caractères fromagers
Nous avons ensuite estimé le progrès génétique réalisé sur les critères fromagers dans la population de taureaux et de vaches de race Montbéliarde de l’entreprise Umotest.
a. Modèle, animaux et génotypages
Une nouvelle évaluation a été réalisée avec le modèle le plus performant, i.e. CTL3 (contrôles élémentaires des trois premières lactations) appliqué à l’ensemble des données, soit 2,6 millions d’observations de 190 261 vaches. Les effets des marqueurs étant estimés directement par la méthode HSSGBLUP, cette analyse fournit les formules de prédiction des valeurs génétiques à partir des génotypes aux marqueurs. Grâce à la disponibilité des génotypages (vrais ou imputés à partir de la puce LD) pour un grand nombre d’animaux de race Montbéliarde de l’entreprise de sélection Umotest (21 171 mâles et 311 761 femelles au total en janvier 2019), ces équations de prédiction peuvent être appliquées aux génotypes des animaux contemporains des individus du pedigree From’MIR, i.e. les taureaux nés entre 2005 et 2018 et les vaches nées entre 2008 et 2018.
b. Evolution des valeurs génétiques des caractères fromagers entre 2005 et 2018
Les courbes d’évolution des moyennes des valeurs génétiques par année de naissance réalisée sur les mâles et les femelles sont présentées sur la figure 6 pour les 11 caractères. On note une amélioration régulière des valeurs génétiques de tous les critères fromagers sur la période 2005-2018, i.e. en moyenne une augmentation des valeurs génétiques de tous les caractères sauf celles du rendement en MSU et des K10/RCT (inverse de la vitesse d’organisation) que l’on cherche à diminuer. Dans la zone AOP Comté, le niveau génétique moyen des taureaux et des vaches de race Montbéliarde génotypés par Umotest est donc aujourd’hui plus élevé qu’en 2005 pour les rendements fromagers et les paramètres de coagulation.
Figure 6. Courbes de l’évolution génétique réalisée par année de naissance des taureaux nés entre 2005 et 2018 et des vaches nées entre 2008 et 2018 pour les 11 caractères fromagers.

Ces résultats sont cohérents avec la sélection pratiquée en race Montbéliarde. Dans cette race, l’index économique laitier donne un poids très important au TP et cet index représente à lui seul 45 % de la pondération économique dans l’index de synthèse global (ISU) de la race. On observe d’ailleurs une réponse forte sur les caséines qui constituent environ 80 % des protéines totales du lait. En race Montbéliarde, la sélection sur le TP a donc dans le même temps amélioré les caractères fromagers et en particulier les paramètres de coagulation qui sont les plus corrélés génétiquement au TP (Sanchez et al., 2018a).
4.3. Études des potentialités de sélection des caractères fromagers
Pour évaluer dans quelle mesure il était possible d’accroître davantage le gain génétique sur les caractères fromagers et estimer l’impact relatif sur les autres caractères, nous avons simulé trois scénarios avec trois objectifs de sélection différents.
i) Le premier scénario correspond à la situation actuelle, la sélection est réalisée sur l’index de synthèse global calculé ainsi :
ISU = 0,45 SYNTLAIT + 0,145 STMA + 0,18 REPRO + 0,05 LGF + 0,05 VTRA + 0,125 MO
avec les index SYNTLAIT = 1,050 (MP + 0,1 MG + 3 TP + 0,5 TB) qui combine les index des matières protéique (MP) et grasse (MP) ainsi que ceux des TP et TB, santé de la mamelle STMA qui combine les index des comptages cellulaires (CEL) et des mammites cliniques (MACL), fertilité REPRO, longévité fonctionnelle LGF, vitesse de traite VTRA et morphologie MO.
ii) Le deuxième scénario ajoute l’index de la teneur en caséines dans le lait (ƩCN) à l’ISU actuel avec les pondérations suivantes :
ISU-COMP = 0,7 ISU + 0,3 ƩCN
iii) Le troisième scénario donne le même poids à l’ISU que précédemment et répartit les 30 % restant à parts égales aux trois caractères fromagers jugés prioritaires par un groupe d’experts fromagers et affineurs de la zone AOP Comté, à savoir le rendement en extrait sec, et la fermeté et la vitesse d’organisation du caillé en modèle pâte pressée cuite :
ISU-FROM = 0,7 ISU + 0,1 (RDT_ES + aPPC - K10/RCTPPC)
Dans ces deux derniers scénarios, le poids accordé aux nouveaux caractères est particulièrement élevé, probablement nettement supérieur à ce qui peut être envisagé en pratique, ils donnent donc une tendance extrême.
Pour chacun des scénarios, nous avons simulé une sélection par troncature en utilisant les valeurs génétiques des taureaux calculées dans le paragraphe précédent pour les caractères fromagers et les index génomiques de l’évaluation officielle pour les autres caractères de l’objectif de sélection. La sélection simulée mime la stratégie de sélection pratiquée par l’entreprise Umotest en race Montbéliarde. Chaque index de synthèse global ISU, ISU-COMP et ISU-FROM a été calculé pour tous les candidats mâles. Pour chaque scénario, les 80 meilleurs taureaux ont été choisis chaque année parmi l’ensemble des candidats disponibles (environ 1600 en moyenne par an, entre 2009 et 2017). Nous avons calculé les réponses à la sélection sur les 11 caractères fromagers, les index élémentaires MP, MG, TP, TB, CEL, MACL et LAIT ainsi que les index de synthèse ISU, STMA, REPRO, MO et VB. L’index élémentaire LAIT correspond au caractère quantité de lait, l’index de synthèse VB est l’index de valeur bouchère et les autres index sont décrits plus haut. Dans chaque scénario et pour chaque index, les réponses à la sélection ont été estimées annuellement en calculant la différentielle de sélection, i.e. la différence entre la moyenne des index des 80 meilleurs taureaux et la moyenne des taureaux candidats. La moyenne des réponses a ensuite été calculée sur les neuf années de la période (2009-2017). Les index étant exprimés en écart-type génétique (ETG), les réponses à la sélection, présentées à la figure 7 pour les trois scénarios, sont également exprimées dans la même unité.
Figure 7. Réponses à la sélection sur ISU, ISU-COMP et ISU-FROM estimées sur les index fromagers et les index des autres caractères (en écart-type génétique).
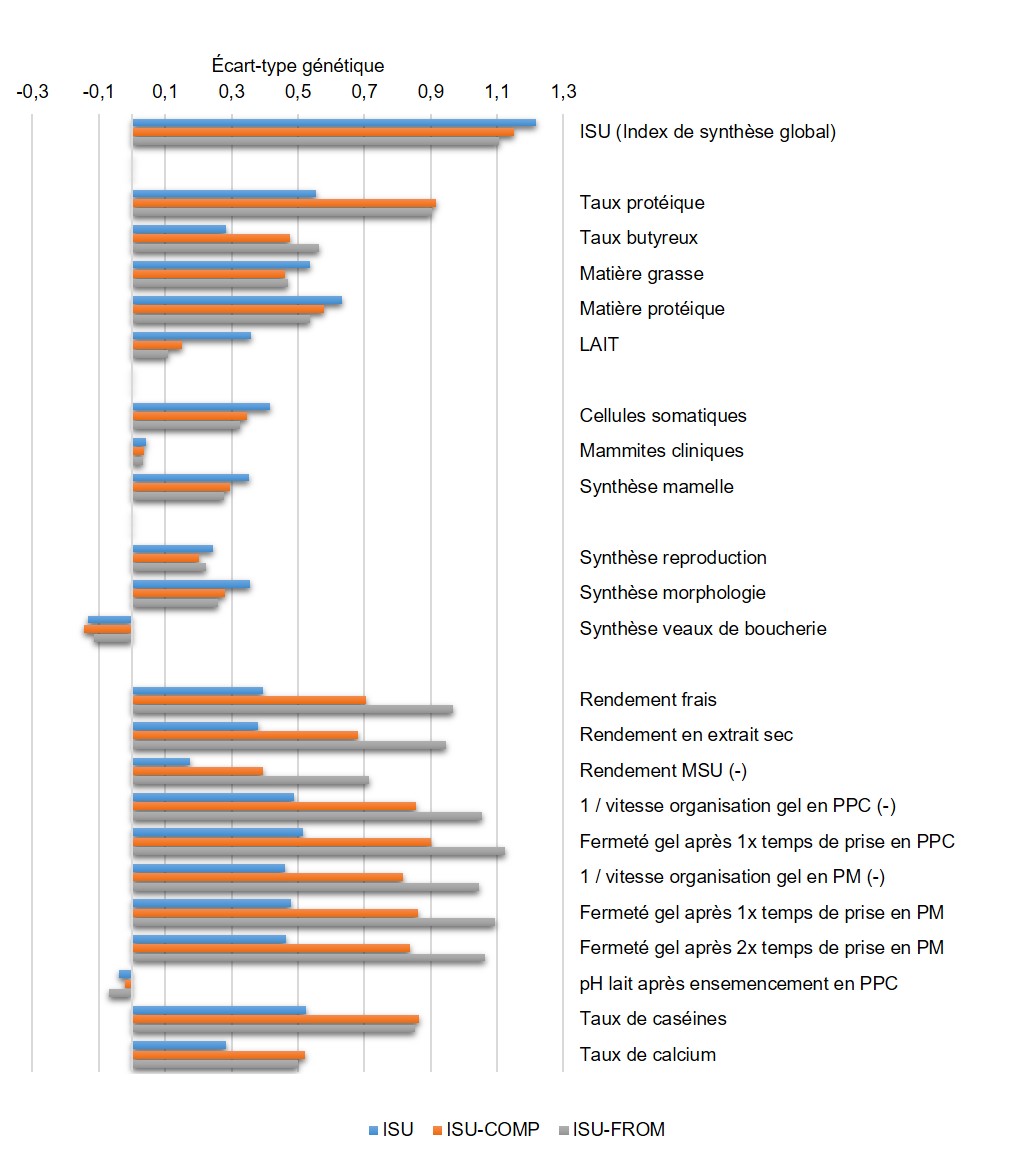
Comme attendu, l’ajout de nouveaux critères dans l’objectif de sélection entraîne des variations de réponse pour tous les index de façon plus ou moins marquée. Pour quasiment tous les caractères, le scénario ISU-COMP entraîne des niveaux de réponse intermédiaires par rapport aux scénarios ISU et ISU-FROM. Pour les caractères autres que les caractères fromagers, comparés à la situation actuelle (ISU), les deux scénarios alternatifs entraînent :
i) une réponse diminuée pour les quantités de matières protéique et grasse et surtout de lait mais une meilleure réponse sur les taux (TP et TB) ;
ii) une réponse légèrement plus faible pour les caractères liés à la santé de la mamelle ;
iii) une réponse quasi-équivalente pour les index de synthèse de fertilité et de valeur bouchère mais une réponse un peu plus faible sur la synthèse morphologie.
Toutefois, les résultats négatifs sont à relativiser dans la mesure où l’ajout des caséines ou des caractères fromagers dans l’objectif de sélection entraîne une diminution du gain de l’ordre de 0,1 ETG pour MP, MG, CEL, MACL, STMA et MO, du fait d’une légère dilution du poids relatif de ces caractères dans l’objectif. Pour le LAIT, cette diminution est plus forte, de l’ordre de 0,2 ETG, et s’explique par la corrélation génétique négative modérée entre les caractères fromagers et le LAIT. Ces valeurs sont relativement limitées, compte tenu du poids élevé accordé au taux de caséines ou aux aptitudes fromagères dans les index alternatifs.
En revanche, le fait de sélectionner les taureaux sur l’ISU-COMP et a fortiori sur l’ISU-FROM entraîne des niveaux de réponse beaucoup plus forts sur l’ensemble des caractères fromagers étudiés dans From’MIR, i.e. tous les rendements fromagers et tous les paramètres de coagulation mesurés sur les fromages de type pâte pressée cuite et pâte molle. Par exemple pour le rendement en extrait sec, par rapport au scénario ISU (+ 0,38 ETG) le gain est nettement amélioré avec le scénario ISU-COMP (+ 0,68 ETG) et a fortiori avec le scénario ISU-FROM (+ 0,95 ETG). Les gains sur les autres caractères fromagers sont du même ordre de grandeur. Les taux de caséines et de calcium dans le lait sont également nettement améliorés de manière à peu près équivalente avec les deux scénarios alternatifs. Pour le taux de calcium par exemple, on passe d’une différentielle de 0,28 ETG avec la sélection sur l’ISU contre 0,50 et 0,52 ETG avec une sélection respectivement sur ISU-FROM et ISU-COMP.
Cette étude montre donc qu’en ajoutant les caractères fromagers dans l’objectif de sélection, il est possible d’obtenir un gain génétique nettement plus conséquent pour les caractères fromagers et pour certains caractères de composition du lait (TP, TB mais aussi caséines et calcium) qu’en sélectionnant sur l’ISU actuel et ce, avec une réduction limitée du progrès génétique des autres caractères.
Conclusion
Le projet From’MIR est le premier de cette ampleur en France pour l’étude des caractères fromagers. Il montre que ces caractères prédits à partir des spectres MIR ont une précision suffisante pour des analyses génétiques. Les rendements fromagers et les paramètres de coagulation sont modérément héritables et ils présentent des corrélations génétiques favorables entre eux et avec les caractères de composition du lait. Nous mettons par ailleurs en évidence un grand nombre de régions du génome qui expliquent une part importante de la variabilité génétique de la composition et de la « fromageabilité » du lait. Dans la plupart de ces régions, des gènes et des variations dans ces gènes sont détectés. Certains gènes sont connus pour leurs effets sur la composition du lait, c’est le cas par exemple des gènes des caséines, DGAT1, PAEP etc. d’autres ont été peu ou pas étudiés jusqu’à présent (SLC37A1, ANKH, SEL1L3, ALPL…). Une approche réseau de gènes fournit une signification biologique à ces résultats via l’identification de voies métaboliques et de gènes régulateurs fonctionnellement liés aux caractères étudiés. Enfin, la population de référence du projet From’MIR (vaches avec phénotypes et génotypes) permet d’estimer des valeurs génomiques précises des caractères fromagers. L’ensemble de ces résultats conduit à la mise en place prochaine d’une évaluation génomique de la « fromageabilité » du lait en race Montbéliarde dans la zone AOP Comté.
Le projet From’MIR s’est focalisé sur les qualités technologiques du lait pour la fabrication de fromages. Or, la qualité des fromages affinés est un critère essentiel, tout spécialement dans un contexte AOP. Cette qualité finale se détermine après une période d’affinage et dépend des conditions de milieu, et tout spécialement de la flore microbienne. Parallèlement à l’évaluation génomique pilote, un observatoire régional de la « fromageabilité » du lait (Observalait) est en cours de création en Franche-Comté. Ses objectifs sont notamment d’entretenir les équations de prédiction avec l’apport des nouvelles données et d’exploiter les spectres MIR pour assurer un suivi en routine des qualités technologiques et organoleptiques du lait à toutes les échelles (individuelle, troupeau et cuve fromagère) jusqu’au fromage affiné (qualité, goût…). À moyen terme, grâce au projet From’MIR, tous les acteurs de la filière (éleveurs, fromagers et affineurs) disposeront donc des outils de phénotypage, de génétique et des conclusions apportées par l’observatoire régional.
Remerciements
Les résultats ont été obtenus dans le cadre du programme From’MIR avec le soutien financier du ministère de l’Agriculture, de l’Agro-alimentaire et de la Forêt, du Centre National Interprofessionnel de l’Économie Laitière (CNIEL), de l’Union Régionale des Fromages d'Appellation d'origine Comtois (URFAC) et de la région Bourgogne Franche-Comté.
Références
- Bittante G., Cipolat-Gotet C., Cecchinato A., 2013. Genetic parameters of different measures of cheese yield and milk nutrient recovery from an individual model cheese-manufacturing process. J. Dairy Sci., 96, 7966-7979.
- Boichard D., Govignon-Gion A., Larroque H., Maroteau C., Palhière I., Tosser-Klop G., Rupp R., Sanchez M.P., Brochard M., 2014. Déterminisme génétique de la composition en acides gras et protéines du lait des ruminants, et potentialités de sélection. In : PhénoFinlait : Phénotypage et génotypage pour la compréhension et la maîtrise de la composition fine du lait. Brochard M., Boichard D., Brunschwig P., Peyraud J.L.(Eds). Dossier, INRA Prod. Anim., 27, 283-298.
- Boichard D., Boussaha M., Capitan A., Rocha D., Hozé C., Sanchez M.P., Tribout T., Letaief R., Croiseau P., Grohs C., Li W., Harland C., Charlier C., Lund M.S., Sahana G., Georges M., Barbier S., Coppieters W., Fritz S., Guldbrandtsen B., 2018. Experience from large scale use of the EuroGenomics custom S.NP chip in cattle. In: 11th WCGALP, Auckland, New Zealand.
- Bonfatti V., Vicario D., Lugo A., Carnier R., 2017. Genetic parameters of measures and population-wide infrared predictions of 92 traits describing the fine composition and technological properties of milk in Italian Simmental cattle. J. Dairy Sci., 100, 5526-5540.
- Caroli A., Chessa S., Erhardt G., 2009. Invited review: Milk protein polymorphisms in cattle: Effect on animal breeding and human nutrition. J. Dairy Sci., 92, 5335-5352.
- CNAOL, 2018. Chiffres clés 2017.
- Corrieu G., Spinnler H., Jomier Y., Picque D., 1988. Automated system to follow up and control the acidification activity of lactic acid starters. French Patent FR 2, 629-712.
- Daetwyler H.D., Capitan A., Pausch H., Stothard P., Van Binsbergen R., Brøndum R.F., Liao X., Djari A., Rodriguez S., Grohs C., Esquerré D., Bouchez O., Rossignol M.N., Klopp C., Rocha D., Fritz S., Eggen A., Bowman P.J., Coote D., Chamberlain A.J., VanTassell C.P., Hulsegge I., Goddard M.E., Guldbrandtsen B., Lund M.S., Veerkamp R.F., Boichard D., Fries R., Hayes B.J., 2014. Whole-genome sequencing of 234 bulls facilitates mapping of monogenic and complex traits in cattle. Nat. Genet., 46, 858-867.
- De Marchi M., Toffanin V., Cassandro M., Penasa M., 2014. Invited review: Mid-infrared spectroscopy as phenotyping tool for milk traits. J. Dairy Sci., 97, 1171-1186.
- El Jabri M., Sanchez M.P., Trossat P., Laithier C., Wolf V., Grosperrin P., Beuvier E., Rolet-Répécaud O., Gavoye S., Gauzere Y., Belysheva O., Notz E., Boichard D., Delacroix-Buchet A., 2019. Comparison of Bayesian and PLS regression methods for mid-infrared prediction of cheese-making properties in Montbéliarde cows. J. Dairy Sci., 102, 6943-6958.
- Fang Z., Visker M., Miranda G., Delacroix-Buchet A., Bovenhuis H., Martin P., 2016. The relationships among bovine alpha(S)-casein phosphorylation isoforms suggest different phosphorylation pathways. J. Dairy Sci., 99, 8168-8177.
- Fernando R., Cheng H., Golden B., Garrick D., 2016. Computational strategies for alternative single-step Bayesian regression models with large numbers of genotyped and non-genotyped animals. Genet. Sel. Evol., 48, 96.
- Fortes M., Reverter A., Zhang Y., Collis E., Nagaraj S., Jonsson N., Prayaga K., Barris W., Hawken R., 2010. Association weight matrix for the genetic dissection of puberty in beef cattle. Proc. Natl. Acad. Sci., USA 107, 13642-13647.
- Gelé M., Minery S., Astruc J.M., Brunschwig P., Ferrand M., Lagriffoul G., Larroque H., Legarto J., Martin P., Miranda G., Palhière I., Trossat P., Brochard M., 2014. Phénotypage et génotypage à grande échelle de la composition fine des laits dans les filières bovine, ovine et caprine. In : PhénoFinlait : Phénotypage et génotypage pour la compréhension et la maîtrise de la composition fine du lait. Brochard M., Boichard D., Brunschwig P., Peyraud J.L. (Eds). INRA Prod. Anim., 27, 255-268.
- Grelet C., Bastin C., Gele M., Daviere J., Johan M., Werner A., Reding R., Pierna J., Colinet F., Dardenne P., Gengler N., Soyeurt H., Dehareng F., 2016. Development of Fourier transform mid-infrared calibrations to predict acetone, beta-hydroxybutyrate, and citrate contents in bovine milk through a European dairy network. J. Dairy Sci., 99, 4816-4825.
- Grosclaude F., 1988. Le polymorphisme génétique des principales lactoprotéines bovines. INRA Prod. Anim.,1, 5-17.
- Hurtaud C., Rulquin H., Delaite M., Verite R., 1995. Prediction of cheese yielding efficiency of individual milk of dairy cows - correlation with coagulation parameters and laboratory curd yield. Ann. Zoot., 44, 385-398.
- Janky R., Verfaillie A., Imrichova H., Van de Sande B., Standaert L., Christiaens V., Hulselmans G., Herten K., Sanchez M., Potier D., Svetlichnyy D., Atak Z., Fiers M., Marine J., Aerts S., 2014. iRegulon: From a gene ist to a gene regulatory network using large motif and track collections. PLoS Comput. Biol., 10.
- Reverter A., Chan E., 2008. Combining partial correlation and an information theory approach to the reversed engineering of gene co-expression networks. Bioinformatics 24, 2491-2497.
- Sanchez M.P., Govignon-Gion A., Croiseau P., Fritz S., Hozé C., Miranda G., Martin P., Barbat-Leterrier A., Letaïef R., Rocha D., Brochard M., Boussaha M., Boichard D., 2017. Within-breed and multi-breed GWAS on imputed whole-genome sequence variants reveal candidate mutations affecting milk protein composition in dairy cattle. Genet. Sel. Evol., 49, 68.
- Sanchez M.P., El Jabri M., Minéry S., Wolf V., Beuvier E., Laithier C., Delacroix-Buchet A., Brochard M., Boichard D., 2018a. Genetic parameters for cheese-making properties and milk composition predicted from mid-infrared spectra in a large dataset of Montbéliarde cows. J. Dairy Sci., 101, 10048-10061.
- Sanchez M.P., Wolf V., El Jabri M., Beuvier E., Rolet-Répécaud O., Gaüzère Y., Minéry S., Brochard M., Michenet A., Taussat S., Barbat-Leterrier A., Delacroix-Buchet A., Laithier C., Fritz S., Boichard D., 2018b. Short communication: Confirmation of candidate causative variants on milk composition and cheesemaking properties in Montbéliarde cows. J. Dairy Sci., 101, 10076-10081.
- Sanchez M.P., Ramayo-Caldas Y., Wolf V., Laithier C., El Jabri M., Michenet A., Boussaha M., Taussat S., Fritz S., Delacroix-Buchet A., Brochard M., Boichard D., 2019. Sequence-based GWAS, network and pathway analyses reveal genes co-associated with milk cheese-making properties and milk composition in Montbéliarde cows. Genet. Sel. Evol., 51, 34.
- Schaeffer L., 2004. Application of random regression models in animal breeding. Livest. Prod. Sci., 86, 35-45.
- Shannon P., Markiel A., Ozier O., Baliga N., Wang J., Ramage D., Amin N., Schwikowski B., Ideker T., 2003. Cytoscape: A software environment for integrated models of biomolecular interaction networks. Genome Res., 13, 2498-2504.
- Wedholm A., Larsen L.B., Lindmark-Månsson H., Karlsson A.H., Andrén A., 2006. Effect of protein composition on the cheese-making properties of milk from individual dairy cows. J. Dairy Sci., 89, 3296-305.
- Yang J., Lee S., Goddard M., Visscher P., 2011. GCTA: A Tool for Genome-wide Complex Trait Analysis. Am. J. Hum. Genet., 88, 76-82.
Résumé
Les aptitudes du lait à la transformation en fromage sont étroitement liées à sa composition. Ces caractères, difficiles à mesurer directement, ont été prédits à partir des spectres dans le moyen infrarouge (MIR) du lait en race Montbéliarde (projet From’MIR). Cet article rassemble les résultats de l’analyse du déterminisme génétique des aptitudes fromagères et de la composition fine du lait, prédites à partir de six millions de spectres MIR de 400 000 vaches. Ces caractères sont modérément à fortement héritables et les corrélations génétiques entre caractères fromagers (rendements et coagulation) et avec la composition du lait (protéines, acides gras et minéraux) sont élevées et favorables. Des analyses d’association (GWAS) et de réseaux de gènes, réalisées à partir des génotypes imputés pour l’ensemble des variations du génome de 20 000 vaches, permettent d’identifier des gènes et des variants candidats ainsi qu’un réseau de 736 gènes impliqués dans des voies métaboliques et des gènes régulateurs fonctionnellement liés à la composition du lait. Enfin, l’estimation de la précision d’une évaluation génomique montre qu’un modèle de type contrôles élémentaires, incluant les variants détectés par les GWAS et présumés causaux, permet de prédire des valeurs génomiques précises. Nous avons par ailleurs simulé une sélection incluant les aptitudes fromagères qui montre les possibilités de sélectionner efficacement les vaches pour qu’elles produisent un lait plus « fromageable », avec un impact limité sur le gain génétique des caractères actuellement sélectionnés. Ces résultats ont conduit à la mise en place d’un prototype d’évaluation génomique en race Montbéliarde dans la zone AOP Comté en 2019
Pièces jointes
Pas de document complémentaire pour cet article##plugins.generic.statArticle.title##
 Vues: 5369
Vues: 5369
Téléchargements
 PDF: 737
PDF: 737
 XML: 183
XML: 183
Articles les plus lus par le même auteur ou la même autrice
- Asma ZENED, Evelyne FORANO, Céline DELBES, Isabelle VERDIER-METZ, Diego MORGAVI, Milka POPOVA, Yuliaxis RAMAYO-CALDAS, Dominique BERGONIER, Annabelle MEYNADIER, Christel MARIE-ETANCELIN, Les microbiotes des ruminants : état des lieux de la recherche et impacts des microbiotes sur les performances et la santé des animaux , INRAE Productions Animales: Vol. 33 No 4 (2020)
- Charlotte DEZETTER, Didier BOICHARD, Nathalie BAREILLE, Bénédicte GRIMARD, Pascale LE MEZEC, Vincent DUCROCQ, Le croisement entre races bovines laitières : intérêts et limites pour des ateliers en race pure Prim’Holstein ? , INRAE Productions Animales: Vol. 32 No 3 (2019)
- Didier BOICHARD, Sandra DOMINIQUE, Marie BÉRODIER, Sébastien FRITZ, Luc DELABY, Corentin FOUÉRÉ, Mekki BOUSSAHA, Anne BARBAT, Utilisation de la semence sexée en production bovine , INRAE Productions Animales: Vol. 37 No 4 (2024)
- Jean-Pierre BIDANEL, Parsaoran SILALAHI, Thierry TRIBOUT, Laurianne CANARIO, Alain DUCOS, Hervé GARREAU, Hélène GILBERT, Catherine LARZUL, Denis MILAN, Juliette RIQUET, Sandrine SCHWOB, Marie-José MERCAT, Claire HASSENFRATZ, Alain BOUQUET, Christophe BAZIN, Joel BIDANEL, Cinquante années d’amélioration génétique du porc en France : bilan et perspectives , INRAE Productions Animales: Vol. 33 No 1 (2020)
- Pascal RAINARD, Gilles FOUCRAS, Didier BOICHARD, Rachel RUPP, Faibles concentrations cellulaires du lait et sensibilité aux mammites des ruminants laitiers , INRAE Productions Animales: Vol. 31 No 4 (2018)
- Gonzalo CANTALAPIEDRA-HIJAR, Cécile MARTIN, Donato ANDUEZA, Milka POPOVA, Diego MORGAVI, Abimael ORTIZ-CHURA, Benoit GRAULET, Isabelle CASSAR-MALEK, Muriel BONNET, Anne DE LA TORRE CAPITAN, Gilles RENAND, Sébastien TAUSSAT, Isabelle ORTIGUES-MARTY, Pierre NOZIÈRE, Mécanismes digestifs et métaboliques associés aux différences inter-individuelles de l’efficience alimentaire chez le bovin allaitant , INRAE Productions Animales: Vol. 36 No 3 (2023): Dossier « Efficience alimentaire des bovins allaitants » & Articles hors dossier
- Laurianne CANARIO, Nicolas BÉDÈRE, Marc VANDEPUTTE, Didier BOICHARD, Jérôme RAOUL, Catherine LARZUL, Quelles génétiques pour les systèmes d’élevages certifiés en agriculture biologique ? , INRAE Productions Animales: Vol. 37 No 2 (2024): Numéro spécial : L’élevage biologique : conditions et potentiel de développement
- Didier BOICHARD , C. GROHS, C. DANCHIN-BURGE, A. CAPITAN, Les anomalies génétiques : définition, origine, transmission et évolution, mode d'action , INRAE Productions Animales: Vol. 29 No 5 (2016)
- Pauline MARTIN, Sébastien TAUSSAT, Aurélie VINET, Frédéric LAUNAY, Dominique DOZIAS, David MAUPETIT, Daniel VILLALBA, Nicolas C. FRIGGENS, Gilles RENAND, Précocité, efficience et résilience des femelles bovines allaitantes , INRAE Productions Animales: Vol. 36 No 3 (2023): Dossier « Efficience alimentaire des bovins allaitants » & Articles hors dossier
- Sébastien TAUSSAT, Clément FOSSAERT, Gonzalo CANTALAPIEDRA-HIJAR, Laurent GRIFFON, Pauline MARTIN, Gilles RENAND, Paramètres génétiques de l’efficience alimentaire et faisabilité d’une sélection en population bovine Charolaise , INRAE Productions Animales: Vol. 36 No 3 (2023): Dossier « Efficience alimentaire des bovins allaitants » & Articles hors dossier

