La Sélection à Parenté Minimum Génomique : principes et résultats pour les deux races caprines laitières principales françaises
Chapeau
La Sélection à Parenté Minimum est une méthode performante d’optimisation combinée du progrès génétique et de la variabilité génétique des reproducteurs. Sa version « génomique » est appliquée depuis 2018 dans les schémas de sélection caprins en France, concomitamment à la mise en œuvre de la sélection génomique. Un bilan de son efficacité est dressé dans cet article.
Introduction
La mise en œuvre de la sélection génomique dans de nombreuses espèces, avec les progrès génétiques supplémentaires qui sont générés, conduit à s'interroger quant à son impact sur la variabilité génétique. Des travaux de simulation (Colleau et al., 2015) montraient que pour une entreprise de sélection française « type » en bovins lait, il est possible de quasi doubler le progrès génétique (+ 70 à + 90 % selon le scénario étudié) en passant de la sélection classique à la sélection génomique sans accélérer l'accroissement de la consanguinité, notamment en augmentant le nombre de taureaux en IA. Dans la réalité, de nombreuses études issues de différents pays (Doekes et al., 2018 ; Forutan et al., 2018 ; Doublet et al., 2019) indiquent que pour une grande race internationale comme la race Holstein, le progrès génétique est toujours largement privilégié aux dépens de la variabilité génétique, particulièrement depuis l'instauration de la sélection génomique. Ce qui à moyen terme pourra augmenter l'impact négatif de la consanguinité sur les caractères en élevage et sur le long terme diminuera le potentiel d'adaptation des animaux, notamment aux conséquences du changement climatique sur les systèmes agricoles.
Des méthodes mathématiques très efficaces, dites « Optimal Contribution Selection » (OCS), ont vu le jour à la fin des années 90 (Meuwissen, 1997 ; Villanueva et Woolliams, 1997) pour gérer conjointement le progrès génétique et la variabilité génétique et donc aider les gestionnaires des schémas de sélection à trouver un compromis. Colleau et al. (2004) ont développé cette idée en minimisant le coefficient de parenté des reproducteurs à progrès génétique fixé et non, comme les auteurs précédents, en maximisant le progrès génétique à parenté fixée. Cette méthode dite de « Sélection à Parenté Minimum » (SPM) a démontré son efficacité dans de nombreuses filières après application sur données réelles (Colleau et Moureaux, 2006 ; Colleau et Tribout, 2008 ; Palhière et al., 2014). Des tests rétrospectifs montrent que la réduction de la parenté moyenne permise par la SPM est de l'ordre de 20 %, pour un progrès génétique identique à celui réellement réalisé par les schémas de sélection. Avec la sélection génomique, ces méthodes devraient être encore plus efficaces puisque les coefficients de parenté tout comme les valeurs génétiques sont estimés de façon plus précise avec l'information des Single Nucleotide Polymorphism (SNP). Utiliser des parentés génomiques est même une nécessité dans cette situation (Sonesson et al., 2012). Si les parentés généalogiques continuent à être utilisées, on sous-estime systématiquement la dégradation de la variabilité génétique, mieux représentée par la variabilité de profils SNP que par celle des pedigrees.
En France, l'amélioration génétique des caprins laitiers repose sur un noyau de sélection d'environ 600 éleveurs et 175 000 femelles pour l'ensemble des deux races principales : Alpine (65 %) et Saanen (35 %). Le schéma de sélection, conduit par l'organisme entreprise de sélection Capgenes, est identique pour les deux races : il s'appuie sur du testage sur descendance (80 boucs évalués sur les performances de leurs filles, par an au total pour les deux races) et sur l'utilisation de l'IA (34 % au sein du noyau de sélection), ce qui lui permet de dégager un progrès génétique annuel important (0,2 écart-type génétique pour la quantité de matière protéique) (Danchin-Burge et al., 2012). Malgré un taux relativement modéré, l'IA a un rôle central au sein du schéma de sélection puisque 90 % des boucs de monte naturelle ont un père d'IA. À partir de 2018, la sélection génomique a été mise en place dans les deux races. Les index sont alors estimés par la méthode du single step GBLUP (Legarra et al., 2009 ; Carillier et al., 2014 ; Carillier-Jacquin et al., 2017) et une étape de pré-sélection est ajoutée, en fermes, pour les jeunes candidats à l'IA sur la base de leur index génomique, préalablement à leur testage sur descendance. Les gains de précision permis par l'évaluation génomique au niveau des index précoces sur information génomique sont bien moindres que ceux observés en bovins laitiers (Carillier et al., 2014 ; Carillier-Jacquin et al., 2017) et ne permettent pas de s'affranchir totalement de l'étape de testage sur descendance. Ainsi, à l'instar de ce qui était appliqué en sélection classique, au moins 30 % des IA doivent être faites avec des jeunes boucs pour assurer leur testage. Toutefois, une modification majeure est opérée au sein du schéma de sélection puisqu'une partie des jeunes boucs d'IA indexés sur information génomique est utilisée conjointement au pool des pères à boucs (PAB) testés sur descendance, afin de réduire l'intervalle de génération. Des travaux de simulation montrent que le gain de progrès génétique attendu dans le cadre d'un schéma de sélection génomique caprin est de l'ordre de + 20 % à + 30 % (Bothorel et Palhière, communication personnelle), ce qui est du même ordre que ce qui a été réalisé dans les races ovines laitières des Pyrénées (+ 16 à + 29 % selon la race, Astruc et al., 2022) mais moindre que dans les 3 grandes races bovines laitières françaises, + 50 %, + 71 %, + 33 %, respectivement en races Montbéliarde, Normande, Holstein (Doublet et al., 2019), où le testage sur descendance n'est plus pratiqué.
La gestion de la variabilité génétique a toujours été une préoccupation majeure pour les gestionnaires des schémas de sélection caprins. Ceci se traduit par un niveau moyen modéré de consanguinité des femelles au contrôle laitier, environ 3 % (Idele, 2021), dans un contexte où la qualité des généalogies est satisfaisante (en moyenne, 10 équivalent-générations remontées en 2020). Quelles que soient les méthodes appliquées, c'est sur l'étape de procréation des boucs d'IA que les efforts ont porté car primordiale comme indiqué plus haut. Un premier bilan en a été effectué en 2014 (Palhière et al., 2014). De 2002 à 2005, une méthode fondée sur une sélection intra famille de PAB a été appliquée. A partir de 2006, Capgenes, en collaboration avec l'INRA, a décidé de mettre en œuvre la SPM pour cette étape. Ainsi, depuis cette date, les accouplements entre PAB et mères à boucs (MAB) sont optimisés une fois par an avec la SPM puis proposés aux éleveurs tout au long de la campagne d'IA. Les résultats sont satisfaisants puisque le rythme d'augmentation de la consanguinité est réduit et le progrès génétique est maintenu constant sur les caractères laitiers malgré l'introduction de nouveaux caractères dans l'objectif de sélection. Avec l'arrivée de la sélection génomique en 2018, la SPM est devenue SPMG (Sélection à Parenté Minimum Génomique), grâce à : i) la prise en compte des parentés génomiques, issues des informations SNP pour les couples d'animaux typés et sinon estimées ; ii) l'utilisation de deux pools de PAB : des PAB testés sur descendance (comme précédemment) et des jeunes boucs non testés sur descendance, ayant des précisions d'index très différentes.
L’objectif de cet article est double :
- présenter de façon détaillée les principes et les méthodes de la SPMG, illustrés par les résultats numériques obtenus lors de l’optimisation des accouplements en 2021 dans les deux races ;
- analyser les variations des progrès génétiques et de la variabilité génétique observées sur les boucs d’IA nés entre 2000 et 2020, de manière à apprécier l’impact réel de la SPM sur le long terme et les premiers effets de la SPMG.
1. La SPMG : principes et méthodes
1.1. Principes généraux et animaux impliqués
La SPM (ou SPMG) est appliquée une fois par an, pour chacune des deux races, à l’étape de procréation des jeunes boucs d’IA. Elle fournit une liste d’accouplements entre PAB et MAB qui seront réalisés tout au long de la campagne chez les différents éleveurs sélectionneurs. En amont de la SPM (ou SPMG), Capgenes indique le nombre d’accouplements souhaités et fournit différentes listes d’animaux. Les effectifs impliqués dans les accouplements réalisés en 2021 sont présentés dans le tableau 1 :
- les MAB candidates : elles correspondent aux chèvres à haut niveau génétique et disponibles pour une mise à la reproduction au cours de la campagne. Le choix est le fruit d’une concertation entre les éleveurs et Capgenes ;
- les PAB candidats : Capgenes fait une sélection de boucs d’IA ayant un haut niveau génétique et/ou une origine génétique peu représentée et/ou des profils génétiques particuliers à certains index élémentaires. Entre 2006 et 2017, lors de la SPM, seuls des boucs testés sur descendance étaient utilisés en tant que PAB (Coefficient de Détermination (CD) des index autour de 0,90). Depuis l’instauration de l’évaluation génomique en 2018, la SPM est devenue SPMG : de jeunes boucs indexés précocément sur information génomique sont alors intégrés (CD autour de 0,55), et sélectionnés sur les mêmes critères ;
- les boucs des 4 millésimes de naissance précédents (4S), issus des accouplements optimisés par la SPM ou la SPMG : les 3 millésimes les plus anciens correspondent à des boucs en cours de testage sur descendance, le millésime le plus récent à des boucs à naître ou juste nés en élevage. Une partie de ces boucs peut être intégrée dans la liste des PAB. L’objectif de la SPM (et SPMG) étant de minimiser la parenté moyenne d’une population globale de futurs boucs reproducteurs, on inclut ces quatre séries de boucs non encore testés sur descendance dans l’optimisation.
Tableau 1. Effectifs d’animaux impliqués dans les accouplements réalisés en 2021.
Alpine |
Saanen |
||
|---|---|---|---|
MAB candidates |
Effectif |
871 |
822 |
Nombre d’élevages |
140 |
92 |
|
PAB candidats |
Effectif Testés sur descendance |
44 |
31 |
Non testés |
107 |
45 |
|
Total |
151 |
76 |
|
Nombre total de doses |
7 550 |
3800 |
|
4S |
Effectif |
415 |
302 |
Animaux du pedigree avec génotype (% femelles) |
2 901 (49 %) |
1 980 (41 %) |
|
Animaux dans le pedigree |
41 557 |
34 678 |
|
MAB : mères à boucs ; PAB : pères à boucs ; 4S : 4 millésimes de boucs non testés sur descendance issus des accouplements lors des 4 années précédentes
Le niveau génétique souhaité pour les produits des accouplements, c’est-à-dire les jeunes boucs à naître, est fixé par Capgenes, parmi plusieurs scénarios possibles (en général 4) et après examen détaillé de toutes les conséquences correspondantes. Il porte sur l’index de synthèse générale correspondant à l’objectif de sélection (appelé ICC) mais peut, en plus, porter sur certains index de synthèse partielle qui le composent (option prévue par la procédure d’optimisation) : l’index de synthèse sur les caractères laitiers (IPC), l’index de synthèse sur les caractères de la morphologie de la mamelle (IMC) ou l’index cellules somatiques (CCS).
Afin de procréer des produits ayant le moins de défauts possibles (voir le détail en paragraphe 1.3.b), une liste de 13 index élémentaires avec un seuil minimum acceptable est également fournie par Capgenes.
Depuis 2018, le passage à la SPMG a nécessité de disposer des génotypages SNP des animaux. En caprins, une seule puce à ADN est disponible sur le marché à ce jour et l’ensemble des animaux est donc génotypé avec la puce 54K Illumina. 46 707 SNP sont utilisés, les mêmes que ceux impliqués en routine dans l’évaluation génomique. En 2021, tous les PAB candidats et tous les jeunes boucs en cours de testage (trois millésimes parmi les 4S) sont génotypés, alors qu’une seule MAB candidate pour chacune des races avait un génotype.
En plus de toutes ces informations, un fichier pedigree de l’ensemble des animaux impliqués est nécessaire dans la SPM tout comme dans la SPMG (tableau 1).
1.2. Méthode de calcul des parentés génomiques
Les parentés génomiques et les consanguinités génomiques sont calculées par la méthode de VanRaden (2008) d'après le degré de similarité de profils SNP entre individus typés et en considérant que les fréquences des deux allèles à chaque SNP sont égales à 0,5 dans la population théorique de base. En partant de l'ensemble des parentés génomiques pour l'ensemble des animaux typés, Legarra et al. (2015) ont proposé des méthodes permettant d'estimer la parenté génomique moyenne (métaparenté) d'une population fictive (dite métafondatrice) d'où proviennent les fondateurs de pedigree. Les valeurs obtenues sont non négligeables (14,6 % en Alpine et 13,4 % en Saanen) et illustrent bien les erreurs que l'on commet en considérant que les fondateurs de pedigrees sont non apparentés et non consanguins.
Dans une première approche, Legarra et al. (2009) estimaient les parentés et consanguinités génomiques concernant les individus non typés, en prenant en compte l'ensemble des parentés génomiques entre typés et l'ensemble des parentés de pedigree qui supposaient les fondateurs comme non apparentés. Ici, on a utilisé une extension de cette approche dans laquelle les parentés de pedigree sont modifiées en partant de parentés entre fondateurs toutes égales à la métaparenté (Legarra et al., 2015). Les calculs numériques correspondants ont été effectués suivant la méthode de Colleau et al. (2017), adaptée au calcul intensif sur grande population. Les parentés génomiques sont donc situées dans l'intervalle (métaparenté, 1) et non dans l'intervalle (0,1) correspondant aux parentés de pedigree, ce qui est susceptible de perturber les sélectionneurs. Perturbation inutile, car ils peuvent réaliser exactement la même qualité de travail en utilisant des parentés génomiques converties grâce à une transformation linéaire. Ici, les deux coefficients de la transformation ont été calculés une fois pour toutes en 2018, au moment de l'instauration de l'évaluation génomique (donc avant tout effet de la sélection génomique sur la variabilité génétique) suivant la méthode proposée par Colleau et al., (2017). Les résultats présentés ici sont en fait des résultats de parenté génomique convertie mais pour simplifier, la référence à la conversion a disparu.
1.3. Méthodes et étapes d’optimisation
La SPMG se déroule en deux étapes distinctes : d’abord le choix des reproducteurs et le calcul de leur taux d’utilisation ; ensuite, le choix des accouplements, c’est-à-dire l’appariement entre les PAB et MAB choisis précédemment (figure 1). Une seule et même méthode, le recuit simulé, est utilisée dans ces deux étapes. L’encadré 1 présente ses principes généraux puis les détails de sa mise en œuvre dans le contexte opérationnel complexe de la SPMG.
Figure 1. Schéma simplifié du fonctionnement général de la SPMG.
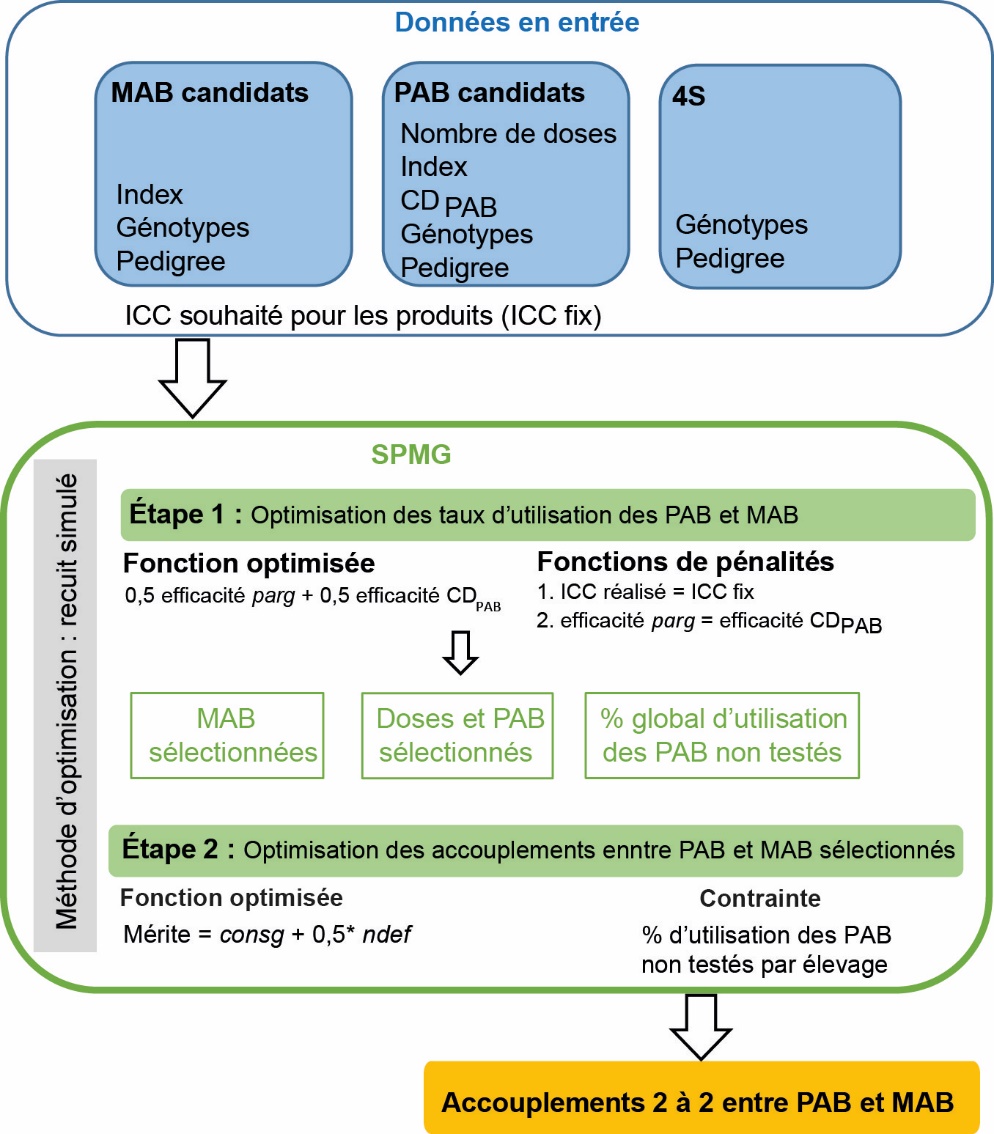
MAB : mères à boucs ; PAB : pères à boucs ; 4S : 4 millésimes de boucs non testés sur descendance issus des accouplements lors des 4 années précédentes ; CDPAB : coefficient de détermination pour l’ICC des PAB ; ICC : index combiné caprin (index de synthèse générale) ; parg : parenté génomique moyenne entre les produits et 4S ; consg : consanguinité génomique ; ndef : nombre moyen de défauts sur 17 index.
Encadré 1. Méthode du recuit simulé.
1. Principes de la méthode du recuit simulé (simulated annealing)
Dans le domaine de la génétique animale, les auteurs font souvent référence à Kirkpatrick et al. (1983) mais en fait, la méthode a été proposée pour la première fois par Metropolis et al. (1953) pour résoudre un problème d'optimisation en physique (avec un très grand nombre de variables).
D'une manière générale, le problème posé consiste à maximiser une fonction de nature quelconque impliquant de très nombreuses variables, elles-mêmes de nature quelconque. Dans ce contexte, la recherche de la configuration optimale du système par une méthode analytique peut s'avérer difficile, voire impossible. Le recuit simulé est donc une recherche empirique de la solution, en quelque sorte un tâtonnement à l'aveugle : c'est une « Monte-Carlo method », pour reprendre la terminologie anglo-saxonne.
Considérons une fonction f dépendant d'une série de variables et qui est maximum pour des valeurs résumées par le terme sol (c'est-à-dire solution). Quand on considère une solution provisoire sol1, la valeur de la fonction est f(sol1). Dans le recuit simulé, on crée ensuite une autre solution provisoire (sol2), en introduisant n'importe où (aléatoirement) une petite modification et l'on calcule la variation de la fonction = f(sol2)-f(sol1). Généralement, mais pas toujours (voir en dessous), on ne l'accepte que si cette variation est positive, auquel cas sol2 devient la nouvelle sol1 et l'on recommence jusqu'à ce que l'on ne trouve plus de meilleure situation. La dernière solution provisoire devient la solution définitive. Colleau et al. (2009), Chapuis et al. (2016) rappellent le détail analytique des algorithmes correspondants. On accepte quelquefois sol2 même quand elle est moins bonne que sol1. Cette acceptation délibérée de « mauvaises » solutions provisoires a pour but d'éviter que l'algorithme trouve un maximum local f(solution locale) inférieur à f(sol, la vraie solution). En effet, en imaginant que f représente une altitude dans un paysage, les fluctuations de f évoquent très souvent une situation de massif montagneux : on peut manquer le sommet le plus élevé en se fourvoyant sur une montagne moins haute.
2. Principes d'une méthode de recuit simulé avec contraintes
Pour le cas où il existe des contraintes, formalisées par des fonctions de contraintes, Colleau et al. (2009) et Chapuis et al. (2016) donnent les principes généraux d'une méthode (CSA, Constrained Simulated Annealing), avec application dans des contextes zootechniques extrêmement différents. Elle est appliquée aussi au cas de la SPMG. Dans cette méthode, les fonctions originelles de contraintes sont transformées en fonctions de pénalités positives ou nulles, tout simplement les carrés des écarts entre valeurs observées et valeurs fixées au niveau des fonctions de contraintes. Les références mentionnées ci-dessus indiquent dans le détail comment il est possible d'obtenir des pénalités nulles dans la solution finale.
De plus, la recherche de l'efficacité opérationnelle conduit aux principes suivants :
- établir l'expression analytique des variations de fonctions quand on passe d'une solution provisoire à la suivante, pour éviter de calculer les fonctions elles-mêmes (sauf à l'initialisation du recuit simulé), ce qui est plus simple et moins long à exécuter ;
- éviter le recours à des fonctions de pénalités quand il est possible de générer des alternatives respectant automatiquement les contraintes considérées, grâce à la mise en œuvre de variables de gestion. Les références mentionnées ci-dessus donnent des exemples de telles variables, dont certaines sont mises en œuvre dans la SPMG.
a. Variables considérées dans l’étape du choix des reproducteurs et du calcul des taux d’utilisation
Il y a un seul accouplement proposé par MAB avec une seule dose de PAB utilisée par accouplement. La somme des contributions des PAB est égale à 0,5, de même que celle des MAB. La contribution d’un PAB particulier est alors 0 ou 0,5 x nombre de doses utilisées/nombre de MAB sélectionnées. La contribution d’une MAB est égale à 0 ou 0,5/ nombre de MAB sélectionnées.
En utilisant ces pondérations, on peut alors calculer l’index moyen des produits et le CD moyen des PAB. La parenté moyenne (en version parg, dans la SPMG,ou parp, dans la SPM) concerne la fenêtre mobile regroupant non seulement les produits mais aussi les quatre séries annuelles antérieures de boucs non testés (4S). C’est un polynôme du second degré impliquant le produit de tous les couples de contributions. On notera que cette fenêtre mobile ne concerne que les voies de transmission père-fils et mère-fils. Normalement, la voie de transmission père-fille intervient aussi dans l’obtention des évolutions asymptotiques annuelles tant du côté des index que du côté de la variabilité génétique.
La procédure d’optimisation considère en fait une variable synthétique appelée « efficacité » qui est la moyenne de deux efficacités élémentaires (sur l’échelle 0-1), concernant l’une la parenté moyenne (parg) et l’autre l’imprécision moyenne des index PAB (1-CDPAB). En effet, ces variables peuvent être considérées comme des variables de nuisance à minimiser, bien entendu sous la contrainte de l’ICC moyen demandé par Capgenes (ICC fix). Le mode de calcul des efficacités élémentaires est donné dans l’encadré 2. Deux fonctions de pénalités sont aussi utilisées, d’une part le carré de la différence ICC observé – ICC objectif (ICC fix) et d’autre part le carré de la différence entre les deux efficacités élémentaires (figure 1). Plusieurs listes de gestion sont considérées et mises à jour en cas d’acceptation de nouvelles solutions :
- liste des MAB utilisées, des MAB non utilisées ;
- liste fixe des PAB « titulaires » des N doses totales (numérotées de 1 à N) ;
- liste des doses utilisées, des doses non utilisées.
Le recuit fonctionne avec deux processus équiprobables de génération des solutions alternatives : i) permutation entre une dose utilisée et une dose non utilisée, ce qui modifie généralement les contributions de deux PAB ; ii) permutation entre une MAB sélectionnée et une MAB non sélectionnée.
Encadré 2. Définition de l’efficacité élémentaire concernant une variable de nuisance.
Soit x la valeur d’une variable de nuisance, à la fin du recuit simulé sous la contrainte ICC moyen = ICC objectif. Toujours sous cette contrainte, le minimum possible est min(x) et le maximum est max(x). Le calcul de min(x) et max(x) est effectué grâce à deux optimisations séparées : minimisation et maximisation. L’efficacité élémentaire pour la variable x est l’expression de x sur une échelle standard 0-1, ce qui permet de comparer des variables de nuisance de nature très différente. Elle est donc égale au rapport (max(x)-x)/(max(x)-min(x)), qui est bien 0 pour max(x) et 1 pour min(x).
Dans la SPMG, il y a deux variables de nuisance, ce qui conduit à effectuer 4 recuits simulés auxiliaires, en préparation du recuit simulé principal.
b. Variables considérées dans l’étape des accouplements
À cette étape, deux variables sont considérées (figure 1) :
- la consanguinité moyenne des produits (en version consg, dans la SPMG, ou consp, dans la SPM) ;
- le nombre moyen de défauts des produits (ndef), sachant qu’il y a défaut pour un index particulier quand celui-ci est inférieur à un seuil. Pour les accouplements 2021, 13 seuils ont été choisis par Capgenes et 4 autres (pour les index ICC, IPC, IMC, CCS) ont été déterminés automatiquement et égaux à la valeur qui correspond aux 20 % inférieurs de tous les accouplements possibles entre PAB et MAB sélectionnés.
La procédure d’optimisation considère, pour chaque accouplement, une variable synthétique, le mérite moyen, qui est une combinaison des moyennes élémentaires sur ces deux variables. Les coefficients de pondération appliqués à chacune des variables sont égaux à – 1 pour consg (ou consp) et – 0,5 pour ndef, après transformation de chaque moyenne en unités d’écart-type respectif, lui-même calculé sur l’ensemble des accouplements possibles MAB sélectionnées x PAB sélectionnés. Les accouplements produits sont donc doublement compensateurs d’abord via les seuils zootechniques et ensuite via la fonction de mérite, conduisant à un équilibre entre divers index zootechniques et la consanguinité.
Normalement, la génération des solutions alternatives est simple : en tirant deux MAB au hasard et en permutant les PAB qui leur sont affectés. Mais on constate que cette procédure, si on la maintient dans le cadre de la SPMG, conduit à une attribution excessive de PAB non testés dans certains élevages de MAB. L’introduction de variables supplémentaires, dont la gestion permet de se déplacer automatiquement dans un espace de solutions satisfaisant pour le sujet considéré, résout ce problème. On détermine donc, élevage par élevage, un nombre de MAB accouplées à des PAB non testés très proche du nombre idéal déterminé par le taux global d’utilisation des PAB non testés. Puis on fait un premier tirage au hasard élevage par élevage des MAB accouplées à des PAB testés ou non testés avec un tirage aléatoire des PAB dans leur catégorie. Ensuite, la génération des solutions alternatives se fait suivant trois processus équiprobables concernant les PAB. Premier processus (intratroupeau) : on tire au hasard un troupeau et on fait l’échange entre un PAB testé et un PAB non testé. Deuxième processus (intertroupeau) : on tire au hasard 2 troupeaux et on échange 2 PAB testés tirés au hasard dans les accouplements concernant ces troupeaux. Troisième processus (intertroupeau) : identique au processus 2 mais avec les PAB non testés. De la sorte, les permutations agissent dans un système autorisant le test de n’importe quelle configuration répondant à la contrainte automatique.
c. Méthode d’optimisation
Une seule et même méthode, le recuit simulé (simulated annealing), est utilisée dans les deux principales étapes. L’annexe (cf. encadré 1) présente ses principes généraux. En ce qui concerne les taux d’utilisation des PAB et des MAB, la méthode vise à maximiser la somme des deux efficacités élémentaires tout en les contraignant à être égales. Il y a donc un effort de réduction équilibrée des deux nuisances (parg ou (1-CDPAB)), car chaque réduction est ressentie comme d’intérêt similaire pour le schéma de sélection. En ce qui concerne les accouplements, on cherche à maximiser le mérite moyen, avec une complication notable : éviter les variations extrêmes entre troupeaux pour le pourcentage d’accouplements avec des boucs non testés en visant une utilisation similaire des boucs non testés par élevage.
2. Réalisation de la SPMG: un cas concret d’application en 2021
2.1. Approche utilisée
L’application de la SPMG porte sur les accouplements réalisés en 2021, pour chacune des deux races caprines laitières françaises, Alpine et Saanen. Les animaux candidats ont été ceux proposés par Capgenes tout comme le nombre d’accouplements souhaités (760 en race Alpine, 700 en race Saanen).
Dans une première approche, les résultats obtenus sont présentés pour 4 scénarios de niveau génétique fixé : celui choisi par Capgenes (ICC de 7,0 en race Alpine et 6,9 en race Saanen ) et une gamme de 3 niveaux génétiques autour du premier, allant jusqu’au niveau génétique maximum permis par la SPMG : i) Alpine : ICC = 6,6/7,4/7,8 ; ii) Saanen : ICC = 6,5/6,7/7,1. La comparaison des animaux candidats aux animaux sélectionnés permet d’évaluer l’efficacité de l’optimisation, en termes de parentés et de niveaux génétiques. Les différentielles de sélection entre candidats et sélectionnés sont exprimées en unités d’écart-type génétique de l’ICC, qui est égal à 3,15 en race Alpine et 3,18 en race Saanen. Les taux d’utilisation des PAB sont également analysés en relation avec la précision des index, qui est très variable selon que ces pères sont testés sur descendance ou non. Une caractérisation des produits est également faite pour évaluer la consanguinité, les niveaux d’index et les défauts pour les différents scénarios. Enfin, une analyse par élevage illustre le pourcentage de MAB retenues ainsi que la proportion accouplée avec des jeunes boucs non testés, proportion qui est optimisée de manière à être très peu variable d’un élevage à l’autre.
Dans une deuxième approche, le scénario 2021 retenu par Capgenes est comparé à un scénario fictif dans lequel l’optimisation serait faite sur les parentés estimées à partir des pedigrees (et non génomiques), les index étant génomiques dans les deux cas. L’objectif est d’illustrer l’intérêt d’utiliser des parentés génomiques.
2.2. Comparaison de 4 scénarios de niveau génétique des accouplements
Dans les scénarios retenus par Capgenes, parmi les MAB candidates, 87 % sont sélectionnées en race Alpine et 85 % en race Saanen. La différentielle de sélection pour l’ICC des MAB sélectionnées est modérée (+ 0,03 écart-type génétique en Alpine, + 0,05 en Saanen) reflétant la faible pression de sélection possible due à un nombre réduit de candidates. En comparaison, la différentielle de sélection des PAB sélectionnés, par rapport aux candidats, est de + 0,09 écart-type génétique en race Alpine et + 0,22 en race Saanen.
a. Taux d’utilisation des PAB
Le tableau 2 montre les résultats obtenus pour les PAB sélectionnés dans les 4 scénarios. Le premier constat est que l'augmentation du niveau génétique souhaité conduit à utiliser un nombre décroissant de PAB et une proportion plus grande de PAB non testés. La conséquence est une baisse du CD moyen des PAB qui atteint 0,66 en race Alpine et 0,71 en race Saanen dans les scénarios extrêmes. L'explication réside dans le fait que les boucs non testés sur descendance présentent, en moyenne, une supériorité génétique notable par rapport aux boucs testés sur descendance (+ 0,36 écart-type génétique sur ICC en race Alpine, + 0,22 en race Saanen), parce qu'ils sont de générations plus récentes et que l'on est en processus de sélection cumulative. On peut noter également que les scénarios choisis par Capgenes ne sont pas ceux réalisant le plus fort progrès génétique mais ceux qui présentent les meilleurs pourcentages d'efficacité d'optimisation et surtout ceux qui utilisent seulement 30 à 40 % de jeunes boucs non testés sur descendance, seuil maximum acceptable par les éleveurs aujourd'hui. Depuis le démarrage de la sélection génomique, le schéma de sélection caprin est dans une phase transitoire entre schéma classique et schéma génomique ce qui explique notamment l'augmentation progressive et contrôlée de l'utilisation de ces jeunes pères à boucs. A terme, cette contrainte sera levée. Dans ces deux scénarios, choisis par Capgenes, le nombre de PAB sélectionnés est très différent selon la race (60 en race Alpine, 37 en race Saanen). Cela résulte, pour la race Alpine, d'un nombre de candidats plus élevé et d'un scénario choisi par Capgenes qui est plus loin du maximum possible. Depuis la SPMG, le nombre de PAB retenus annuellement est en moyenne de 50 pour chaque race, soit le double de ce qui était fait avec la SPM (Palhière et al., 2014).
Tableau 2. Comparaison des effectifs et catégories de PAB sélectionnés selon 4 scénarios d’optimisation faits avec la SPMG, en comparaison des PAB candidats, pour les races Alpine et Saanen.
Race Alpine |
Scénario |
||||
Candidats |
ICC=6,6 |
ICC=7,0 |
ICC=7,4 |
ICC=7,7 |
|
Efficacité d’optimisation |
84 % |
85 % |
82 % |
79 % |
|
Nombre PAB (% doses) testés |
44 (29 %) |
30 (82 %) |
24 (61 %) |
9 (40 %) |
5 (31 %) |
non testés |
107 (71 %) |
18 (18 %) |
36 (39 %) |
44 (60 %) |
28 (69 %) |
total |
151 |
48 |
60 |
37 |
33 |
CDPAB testés |
0,90 |
0,92 |
0,90 |
0,90 |
0,90 |
non testés |
0,57 |
0,60 |
0,57 |
0,56 |
0,54 |
total |
0,67 |
0,86 |
0,77 |
0,69 |
0,66 |
Race Saanen |
Scénario |
||||
Candidats |
ICC=6,5 |
ICC=6,7 |
ICC=6,9 |
ICC=7,1 |
|
Efficacité d’optimisation |
85 % |
86 % |
87 % |
82 % |
|
Nombre PAB (% doses) testés |
31 (41 %) |
22 (77 %) |
18 (72 %) |
14 (66 %) |
7 (48 %) |
non testés |
45 (59 %) |
18 (23 %) |
22 (28 %) |
23 (34 %) |
23 (52 %) |
total |
76 |
40 |
40 |
37 |
30 |
CDPAB testés |
0,90 |
0,89 |
0,89 |
0,89 |
0,89 |
non testés |
0,56 |
0,57 |
0,56 |
0,54 |
0,54 |
total |
0,70 |
0,81 |
0,79 |
0,77 |
0,71 |
Le scénario retenu par Capgenes est en gras ; PAB : pères à boucs ; CDPAB : coefficient de détermination pour l’ICC des PAB ; ICC : index combiné caprin (index de synthèse générale)
La figure 2 illustre la contribution des PAB dans les deux scénarios choisis par Capgenes, en distinguant les PAB testés sur descendance et les non testés. Au minimum, un PAB contribue à 7 accouplements et au maximum à 50 conformément aux exigences de Capgenes (bornes paramétrables). Même si la gamme de contribution des PAB est étendue, on remarque qu'une majorité de PAB contribue peu (≤ 20 doses chacun, soit ≤ 3 % des accouplements), notamment en race Alpine. Le taux d'utilisation est très dépendant de la catégorie du PAB : en moyenne, un PAB testé est accouplé avec 19 MAB en race Alpine et 33 en race Saanen, alors qu'un PAB non testé est accouplé avec 8 et 10 MAB respectivement (soit 2,4 et 3,3 fois moins, respectivement). Le fait que les PAB non testés sur descendance aient des index moins précis conduit donc à une utilisation plus parcimonieuse, malgré leur niveau génétique supérieur. La contribution très déséquilibrée entre PAB est une spécificité de la SPM, et encore plus de la SPMG, comparée à d'autres méthodes de gestion de la variabilité génétique. En effet, en l'absence d'information précise sur le niveau de parenté des reproducteurs et de gestion optimisée, il est recommandé de disposer d'une descendance la plus équilibrée possible par reproducteur, en particulier pour les mâles d'IA. Les mâles les plus et les moins originaux n'étant alors pas repérés, il est plus sage de ne pas faire une utilisation différenciée. C'est ce qui était appliqué avant 2006 pour les caprins (Palhière et al., 2014) et ce qui continue d'être appliqué dans les schémas ovins laitiers (Buisson et al., 2018). À l'inverse, avec la SPMG, les mâles trop apparentés à la population sont peu ou pas utilisés et vice versa.
Figure 2. Répartition des PAB testés sur descendance et non testés selon leur contribution (en nombre de doses) aux accouplements 2021 dans le scénario choisi par Capgenes, pour les races Alpine et Saanen.

Dans le cadre de la SPMG, on observe que le taux d’utilisation des PAB (ici, le nombre de doses) est corrélé à leur ICC : la corrélation est égale à 0,57 en race Alpine et 0,47 en race Saanen pour les PAB testés ; elle est égale à 0,47 en race Alpine et 0,78 en race Saanen pour les PAB non testés. Ce taux d’utilisation est corrélé à peu près de la même manière (en valeur absolue) à leur parenté moyenne avec les 4 séries annuelles antérieures de jeunes boucs : les corrélations s’élèvent à – 0,52 en race Alpine et – 0,50 en race Saanen pour les PAB testés tandis qu’elles sont égales à – 0,61 en race Alpine et – 0,46 en race Saanen pour les PAB non testés. Ceci illustre bien l’intérêt de la SPMG, qui permet de choisir des reproducteurs satisfaisant un compromis équilibré entre progrès génétique et variabilité génétique.
b. Caractéristiques des accouplements
Les résultats de la SPMG sur les produits issus des accouplements sont présentés au tableau 3 pour les 4 scénarios de chaque race. Une augmentation du niveau génétique souhaité induit logiquement une augmentation de la consanguinité moyenne et de la parenté moyenne des produits. Mais l’évolution de ces dernières n’est pas linéaire, leur niveau moyen augmentant moins vite que l’ICC. Il est donc primordial, en pratique, de faire tourner plusieurs scénarios afin de trouver celui offrant le meilleur compromis. On peut noter que, pour les deux races, le scénario maximum sur ICC (ICC=7,7 en Alpine et ICC=7,1 en Saanen) conduit à des niveaux de parenté et de consanguinité moyennes très élevés, qui décrochent par rapport aux scénarios précédents. Le gain d’ICC est dans ce cas très pénalisé par une perte de variabilité génétique. Il est donc compréhensible que Capgenes n’ait pas choisi ces scénarios. Si on s’attache uniquement à ces critères, le choix de Capgenes semble le meilleur compromis en race Saanen. En race Alpine, le scénario choisi par Capgenes favorise la diversité génétique aux dépens du niveau génétique des produits. Le scénario avec ICC=7,4 aurait permis un progrès génétique supérieur tout en maintenant la consanguinité et la parenté moyennes à des niveaux satisfaisants. Mais, avec 60 % d’utilisation de jeunes boucs non testés (tableau 2), ce scénario n’était pas éligible.
L’optimisation des accouplements vise également (en plus de réduire la consanguinité) à limiter le nombre de défauts chez les produits, un défaut correspondant à un index en dessous d’un seuil. Pour les accouplements 2021, 17 index étaient concernés par un seuil minimum (13 fixés par Capgenes et 4 calculés automatiquement, cf 1.3.b). Le tableau 3 montre que pour la majorité des scénarios, le nombre moyen de défauts est très faible, et n’excède jamais 3 pour un produit donné. Il diminue au fur et à mesure que le niveau ICC des produits augmente, du fait que les PAB sélectionnés ont non seulement un niveau ICC de plus en plus élevé mais aussi des index élémentaires plus élevés. Le nombre de défauts est donc diminué. Si on compare à la situation avant optimisation, c’est-à-dire dans l’ensemble des accouplements possibles entre PAB et MAB sélectionnés, le pourcentage d’accouplements à défauts sur l’IPC (index de synthèse pour les caractères laitiers) passe, par exemple, de 30 % avant optimisation à 8 % après optimisation en race Alpine et de 13 à 0,3 % en race Saanen, pour les deux scénarios choisis par Capgenes. Cette étape d’optimisation des accouplements au sein de la SPMG est donc très efficace pour procréer des mâles d’IA ayant des profils d’index équilibrés.
Tableau 3. Indicateurs de variabilité génétique et nombre de défauts des produits issus des accouplements selon 4 scénarios d’optimisation faite avec la SPMG pour les races Alpine et Saanen.
Race Alpine |
||||
|---|---|---|---|---|
Scénario |
ICC=6,6 |
ICC=7,0 |
ICC=7,4 |
ICC=7,7 |
ICC moyenne |
6,60 |
7,0 |
7,40 |
7,69 |
+ 6 % |
+ 12 % |
+ 17 % |
||
Consanguinité (%) moyenne |
2,94 |
2,96 |
3,02 |
3,19 |
+ 1 % |
+ 3 % |
+ 9 % |
||
Parenté moyenne 2 à 2 (%) |
4,70 |
4,74 |
4,94 |
5,34 |
+ 1 % |
+ 5 % |
+ 14 % |
||
Parenté moyenne avec les boucs des 4 millésimes précédents (%) |
4,62 |
4,70 |
4,82 |
4,98 |
ndef moyenne |
0,50 |
0,19 |
0,04 |
0,02 |
Race Saanen |
||||
Scénario |
ICC=6,5 |
ICC=6,7 |
ICC=6,9 |
ICC=7,1 |
ICC moyenne |
6,50 |
6,70 |
6,90 |
7,10 |
+ 3 % |
+ 6 % |
+ 9 % |
||
Consanguinité (%) moyenne |
3,61 |
3,54 |
3,58 |
3,68 |
– 2 % |
– 1 % |
+ 2 % |
||
Parenté moyenne 2 à 2 (%) |
5,47 |
5,47 |
5,59 |
5,88 |
0 % |
+ 2 % |
+ 7 % |
||
Parenté moyenne avec les boucs des 4 millésimes précédents (%) |
5,24 |
5,27 |
5,35 |
5,52 |
ndef moyenne |
0,13 |
0,12 |
0,09 |
0,06 |
Le scénario retenu par Capgenes est en gras ; ICC : index combiné caprin (index de synthèse générale), exprimé en unité d’index. La consanguinité et les parentés sont génomiques. Les pourcentages d’évolution (en italique) sont calculés pour la moyenne (en comparaison au scénario ayant l’ICC le plus bas). ndef : nombre moyen de défauts sur 17 index
Le tableau 4 illustre l’efficacité globale de la SPMG (étape 1 et étape 2) en comparant les résultats obtenus à partir de tous les accouplements possibles entre PAB et MAB candidats aux résultats du scénario retenu par Capgenes. La situation « sans optimisation » peut s’apparenter à une situation de panmixie car alors tous les PAB et MAB candidats contribuent à la génération suivante et sont accouplés sans contrainte. Il ressort que le gain permis par la SPMG est retrouvé à la fois sur le niveau génétique et sur la consanguinité des produits. L’effet conjugué des 2 étapes d’optimisation conduit à une réduction drastique de 2,01 points de consanguinité des produits en race Alpine et 1,94 point en race Saanen, soit une diminution de 35 à 40 % par rapport au scénario sans optimisation. En parallèle, le niveau génétique augmente de 0,21 unité d’ICC en race Alpine et 0,43 en race Saanen, correspondant à une différence de 0,07 et 0,14 écart-type génétique, respectivement.
Tableau 4. Comparaison des valeurs moyennes de l’ICC et de la consanguinité des produits dans une situation sans optimisation (panmixie) et avec optimisation (pour le scénario retenu par Capgenes) pour les races Alpine et Saanen.
Alpine |
Saanen |
|||||
|---|---|---|---|---|---|---|
Sans |
Avec |
δ |
Sans |
Avec |
δ |
|
ICC |
6,79 |
7,00 |
+ 0,21 |
6,47 |
6,90 |
+ 0,43 |
Consanguinité (%) |
4,97 |
2,96 |
– 2,01 |
5,52 |
3,58 |
– 1,94 |
ICC : index combiné caprin (index de synthèse générale), exprimé en unité d’index ; δ correspond à la différence entre les résultats avec optimisation et sans optimisation.
c. Bilan par élevage impliqué dans les accouplements
Un bilan de la SPMG à l’échelle des troupeaux impliqués est présenté dans le tableau 5. Les troupeaux avec des MAB sélectionnées sont au nombre de 135 en race Alpine et 89 en race Saanen, soient 96 à 97 % des troupeaux avec des MAB candidates. Parmi les 600 élevages du noyau de sélection total des deux races, on constate donc que seul un tiers d’entre eux (37 %) participe annuellement aux accouplements programmés. Le nombre de MAB sélectionnées par élevage est variable (1 à 30), et plus élevé en race Saanen qu’en race Alpine, en moyenne. Ceci reflète les différences de taille de troupeau. Comme expliqué au paragraphe 1.3.c, le pourcentage de PAB non testés par troupeau est optimisé de façon à homogénéiser les risques liés à leur CD nettement plus faible. L’idée est de se rapprocher de la situation du schéma classique, dans lequel tous les élevages sélectionneurs avaient « contractuellement » 30 % de doses de boucs en testage sur descendance. Afin d’illustrer l’efficacité de la SPMG, on se concentre sur les élevages ayant au moins 4 MAB accouplées, c’est-à-dire 74 élevages en race Alpine et 64 en Saanen (tableau 4). Dans ces élevages, le pourcentage de PAB non testés varie de 33 à 50 % en race Alpine et de 25 à 40 % en race Saanen. De plus, seuls 17 élevages en race Alpine ont 45 % de PAB non testés ou plus et 11 élevages en race Saanen ont 40 % de PAB non testés ou plus. La SPMG permet donc une répartition équilibrée des PAB non testés par élevage ce qui garantit une meilleure acceptation des accouplements proposés, par les éleveurs.
Tableau 5. Statistiques par élevage ayant des MAB sélectionnées après optimisation avec la SPMG pour le scénario Capgenes en races Alpine et Saanen.
Alpine |
Saanen |
|
|---|---|---|
Nombre de troupeaux sélectionnés |
135 |
89 |
Nombre de MAB accouplées par troupeau moyenne |
5,63 |
7,87 |
Nombre (et %) de troupeaux avec >= 4 MAB sélectionnées |
74 (55 %) |
64 (72 %) |
% de doses de PAB non testés par troupeau* moyenne |
42 % |
33 % |
Nombre (et %) de troupeaux avec >=35 % de PAB non testés |
23 élevages |
|
>=40 % de PAB non testés |
52 élevages |
11 élevages |
>=45 % de PAB non testés |
17 élevages |
0 élevage |
>= 50 % de PAB non testés |
17 élevages |
0 élevage |
*pour les 74 troupeaux avec au moins 4 MAB sélectionnées en race Alpine ; 64 troupeaux en race Saanen. MAB : mères à boucs ; PAB : pères à boucs
2.3. Intérêt de l’utilisation des parentés génomiques
Le tableau 6 présente les résultats de parentés et consanguinités obtenus dans le cas d’un scénario fictif dans lequel l’optimisation serait faite à partir de parentés estimées sur la base des pedigree (et non des génotypages), appelé « optimisation pedigree ». Ils concernent les deux types de parentés et consanguinités (estimées à partir des pedigrees ou des informations génomiques) et sont comparés aux résultats obtenus dans le scénario choisi par Capgenes, appelé « optimisation génomique ». Les index sont génomiques dans les deux scénarios et les niveaux génétiques des produits sur l’ICC sont identiques. Les valeurs « vraies » de consanguinité et de parenté ne sont pas connues mais on fait l’hypothèse que les critères estimés avec les informations génomiques sont très proches de celles-ci. Du tableau 6, deux phénomènes ressortent :
- « l'optimisation pedigree », c'est-à-dire l'utilisation de critères estimés à partir des pedigrees, conduit à une sous-estimation de la consanguinité et de la parenté réelles des reproducteurs, dans un contexte de sélection génomique. En effet, la consanguinité généalogique moyenne des produits est de 2,78 % en race Alpine et 3,50 % en race Saanen alors qu'elle est égale à 3,39% et 4,07 %, respectivement, avec l'information génomique. La sous-estimation varie selon le critère considéré mais semble plus marquée pour la consanguinité moyenne des produits (22 % en race Alpine et 16 % en race Saanen). Ces résultats sont en accord avec les travaux de simulation de Sonesson et al. (2012), qui ont décrit pour la première fois ce phénomène. Alors que dans notre cas une seule génération de sélection est considérée, Sonesson et al. (2012) ont estimé l'impact après 10 générations successives. Ils montrent que la sous-estimation du taux d'augmentation de la consanguinité estimé par les pedigrees s'accroît au fil des générations, pour atteindre 292 % à la dixième génération.
- « l’optimisation génomique » donne les meilleurs résultats quel que soit le critère génomique. Par exemple, la consanguinité génomique moyenne des produits est de 2,96 % en race Alpine et 3,58 % en race Saanen avec une « optimisation génomique », soit – 13 et – 12 %, respectivement, par rapport à une « optimisation pedigree ».
L’ensemble de ces résultats démontre donc qu’en cas d’évaluation génomique l’optimisation doit intégrer des parentés et consanguinités génomiques.
Tableau 6. Comparaison des valeurs moyennes de consanguinité et de parentés des produits, estimées à partir des pedigrees seuls (critères P) ou avec les génotypages (critères G), selon deux modalités d’optimisation, à même niveau génétique.
Race |
Alpine |
Saanen |
||||
|---|---|---|---|---|---|---|
Critère |
P |
G |
Différence G-P |
P |
G |
Différence G-P |
Optimisation pedigree |
||||||
Consanguinité produits |
2,78 |
3,39 |
+ 22 % |
3,50 |
4,07 |
+ 16 % |
Parenté produits |
4,22 |
4,84 |
+ 15 % |
5,20 |
5,67 |
+ 9 % |
Parenté produits x 4S |
4,13 |
4,74 |
+ 15 % |
4,94 |
5,39 |
+ 9 % |
Optimisation génomique |
||||||
Consanguinité produits |
2,95 |
2,96 |
0 % |
3,73 |
3,58 |
– 4 % |
Parenté produits |
4,27 |
4,74 |
+ 11 % |
5,26 |
5,59 |
+ 6 % |
Parenté produits x 4S |
4,16 |
4,70 |
+ 13 % |
4,97 |
5,35 |
+ 8 % |
4S correspond aux 4 millésimes de boucs non testés sur descendance issus des accouplements lors des 4 années précédentes. Optimisation pedigree : utilise les parentés pedigree. Optimisation génomique : utilise les parentés génomiques. La différence G-P est calculée ainsi : (critère génomique – critère pedigree) / critère pedigree. Fait pour les scénarios choisis par Capgenes.
3. Étude des mâles d’IA nés entre 2000 et 2020
3.1. Animaux et critères étudiés
L’étude des mâles d’IA par millésime de naissance, entre 2000 et 2020, permet d’évaluer l’impact final de la SPM et d’estimer les premiers effets de la SPMG. Les mâles d’IA correspondent aux mâles mis en testage sur descendance en sélection classique (millésimes 2000 à 2018) tout comme en sélection génomique (millésimes 2019-2020). Tous sont issus des accouplements raisonnés. Ils reflètent donc bien ce qui a été prévu lors des optimisations, d’autant que 93 % des accouplements proposés dans le cadre de la SPM sont respectés par les éleveurs et que Capgenes essaie de respecter les contributions initiales de chaque père, déterminées par la SPM (ou la SPMG), au moment de l’entrée des jeunes mâles en centre. Le nombre de mâles d’IA considéré est de 959 en race Alpine (soit 46 par millésime en moyenne) et 694 en race Saanen (soit 33 par millésime en moyenne). Au moment de l’étude, les accouplements destinés à procréer les millésimes 2021 et 2022 sont réalisés alors que le choix des mâles pour le testage sur descendance n’est pas encore connu. Pour ces deux millésimes, les résultats obtenus au niveau des accouplements seront pris en compte afin d’essayer de se projeter. L’ensemble des millésimes étudiés peut être divisé en trois périodes : une période « sans SPM » (millésimes 2000 à 2006), une période « SPM » (millésimes 2007 à 2018) et une période « SPMG » (millésimes 2019 à 2022). Des résultats moyens par période ont été estimés afin d’évaluer les changements occasionnés par la SPM d’abord, puis la SPMG. Un calcul sur l’ensemble de la période 2000-2020 est également présenté. Les critères analysés concernent :
- le progrès génétique : 4 index sont considérés : ICC, IPC, IMC, CCS. Ils sont exprimés en unité d’écart-type génétique afin d’avoir une comparaison plus facile entre index et entre races. Les index sont issus de l’évaluation génétique officielle de juin 2021, réalisée par GenEval, association en charge des évaluations génétiques en ruminants. Pour chaque index, des pentes de régression intra-période ont été calculées afin d’évaluer le progrès génétique annuel réalisé ;
- la variabilité génétique : la consanguinité moyenne et la parenté moyenne avec l’ensemble des mâles du même millésime sont estimées pour chacun des mâles d’IA, à partir des informations génomiques (cf méthode paragraphe 1.3.b.) ou des informations généalogiques. Le nombre d’équivalent générations connues dans le pedigree est également calculé afin de vérifier l’impact de la connaissance des généalogies sur les critères issus des pedigrees. Pour chacun des critères, des pentes ont été estimées intra période. Dans notre cas, le calcul des pentes annuelles peut être perturbé par un phénomène de transition de pentes. C’est manifeste pour la consanguinité et la parenté quand on passe de la période sans SPM à la période SPM, où elles diminuent momentanément. Et il est très probable (il est encore trop tôt pour statuer) qu’il se manifestera aussi pour les mêmes variables lors du passage de la période SPM à la période SPMG. Les pentes sont ainsi calculées en ne tenant pas compte de la première année correspondant au changement de régime. Pour les pentes concernant l’ICC, le phénomène n’est pas observé parce que c’est Capgenes qui fixe l’évolution en quelque sorte.
3.2. Bilan global de la diversité génétique et du progrès génétique par période
Le tableau 7 présente les pentes de régression selon l’année de naissance des boucs d’IA pour différents critères de variabilité génétique et les principaux index. Le nombre moyen d’équivalent générations connues pour les mâles d’IA nés entre 2000 et 2020 est de 9,4 en race Alpine et 8,8 en race Saanen, indiquant que les critères de variabilité génétique estimés à partir des pedigrees sont fiables.
Tableau 7. Pentes annuelles d’évolution des critères de variabilité génétique (en %) et d’index (en unité d’écart-type génétique) pour trois périodes de naissance des mâles d’IA et au total sur la période 2000-2022.
Période |
sans SPM |
SPM |
SPMG |
Total |
|---|---|---|---|---|
Race Alpine |
||||
Consanguinité G |
0,30 |
0,14 |
– 0,08 |
0,07 |
Parenté G |
0,13 |
0,01 |
– 0,19 |
0,05 |
Consanguinité P |
0,10 |
0,09 |
0,06 |
0,04 |
Parenté P |
0,01 |
– 0,03 |
– 0,13 |
0,03 |
ICC |
0,14 |
0,14 |
0,19 |
0,17 |
IPC |
0,14 |
0,12 |
0,16 |
0,14 |
IMC |
– 0,01 |
0,07 |
0,03 |
0,06 |
CCS |
– 0,11 |
0,08 |
0,06 |
0,06 |
Race Saanen |
||||
Consanguinité G |
0,22 |
0,17 |
0,07 |
0,05 |
Parenté G |
0,13 |
0,01 |
– 0,06 |
0,04 |
Consanguinité P |
0,08 |
0,12 |
0,03 |
0,06 |
Parenté P |
0,03 |
0,01 |
– 0,07 |
0,03 |
ICC |
0,17 |
0,15 |
0,21 |
0,17 |
IPC |
0,19 |
0,14 |
0,18 |
0,15 |
IMC |
– 0,05 |
0,00 |
0,07 |
0,02 |
CCS |
– 0,04 |
0,06 |
– 0,04 |
0,06 |
La période sans SPM correspond aux millésimes 2000 à 2006, SPM : 2007-2018, SPMG : 2019-2022. La lettre « G » correspond aux critères de consanguinité et de parenté génomiques. la lettre « P » correspond aux critères de consanguinité et parenté pedigree. ICC est l’index de synthèse globale correspondant à l’objectif de sélection. IPC est l’index de synthèse pour les caractères laitiers. IMC est l’index de synthèse pour les caractères de morphologie. CCS est l’index cellules somatiques.
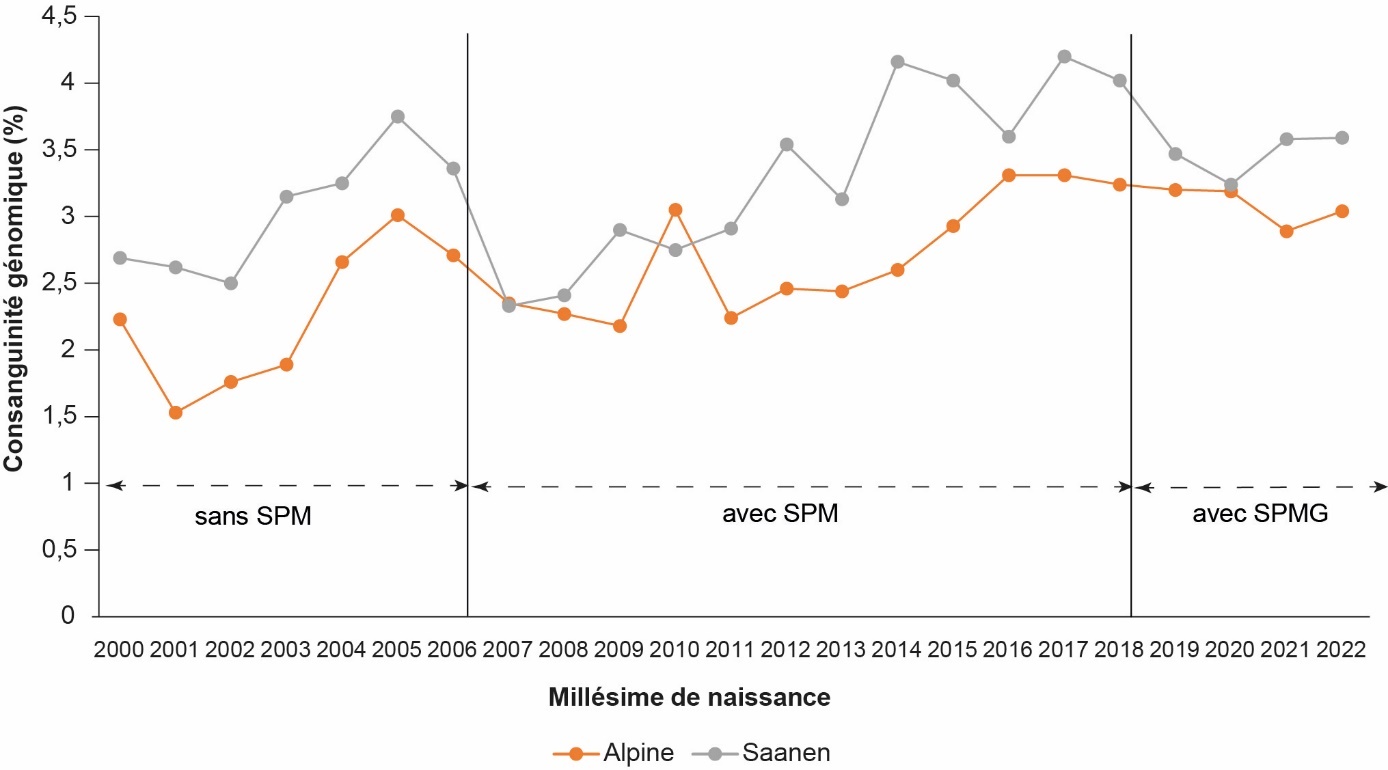
Depuis la mise en place de la SPM, l'augmentation de la consanguinité génomique est fortement ralentie dans les deux races : elle est passée de 0,30 à 0,14 %/an en race Alpine (– 52 % entre période sans SPM et SPM) et de 0,22 à 0,17 %/an en race Saanen (– 20 %). Le rythme d'augmentation est encore moindre pendant la période SPMG mais il est encore un peu tôt pour conclure sur l'effet de la SPMG compte tenu du peu de recul que l'on a. Même s'il fluctue selon les millésimes (figure 3), le taux d'augmentation de la consanguinité est en moyenne très modéré. Il s'élève à 0,05 %/an en race Saanen et 0,07 %/an en race Alpine sur l'ensemble de la période 2000-2022, soit une augmentation par génération de 0,21 % en race Saanen et 0,26 % en race Alpine. Ces résultats sont très proches de ceux obtenus en ovins laitiers dans le cadre d'un schéma classique (Buisson et al., 2018), notamment de la race Lacaune (0,29 %/génération). Mais ils sont nettement inférieurs à ceux présentés par Doublet et al. (2019) pour les 3 races bovines laitières françaises principales, le maximum étant observé pour la race Holstein avec sélection génomique (1,39 %/génération).
3.3. Évolution de la consanguinité des mâles d’IA au cours du temps
Le niveau de consanguinité génomique est également très satisfaisant puisqu'il atteint 3,59 % en race Saanen et 3,04 % en race Alpine, pour les mâles d'IA nés en 2022 (figure 3). Ces chiffres sont très proches de ceux estimés par l'observatoire de la variabilité génétique des races caprines (idele, 2021) sur les femelles au contrôle laitier nées entre 2017 et 2020. En revanche, ils sont très inférieurs à ceux observés pour les taureaux nés en 2015 en race Normande et Montbéliarde (de l'ordre de 6 %) et en race Holstein (de l'ordre de 7 %) (Doublet et al., 2019). La parenté génomique moyenne des mâles intra-millésime montre la même tendance d'évolution au cours des trois périodes que la consanguinité. Ces éléments confirment donc que l'application de la SPM a conduit à une amélioration des critères de variabilité génétique dans les 2 races.
Figure 3. Évolution de la consanguinité génomique moyenne par millésime de naissance des mâles d’IA en race Alpine et Saanen.
3.4. Évolution du niveau génétique des mâles d’IA au cours du temps
L'évolution de l'index de synthèse global (ICC) par millésime de naissance est présentée en figure 4. Le progrès génétique réalisé est relativement constant sur la période et identique dans les deux races. Pour la période 2000-2022, le progrès génétique annuel est de 0,17 unité d'écart-type génétique dans les deux races. En comparaison, les schémas des races ovines laitières généraient des progrès génétiques annuels assez équivalents en sélection classique : de 0,23 écart-type génétique en race Lacaune à 0,16 en race Basco-Béarnaise, pour l'index de synthèse (Astruc et al., 2016). En bovins lait, des travaux de simulation montraient que pour une entreprise de sélection française « type » passée à la sélection génomique, le progrès génétique annuel espéré pourrait atteindre 0,47 écart-type génétique, dans le scénario maximum (Colleau et al., 2015). Si on regarde les progrès génétiques par période (tableau 7), il apparait que la mise en place de la SPM n'a conduit à aucun changement en race Alpine alors que le progrès génétique annuel a légèrement été réduit en race Saanen (– 14 % entre période sans SPM et avec SPM). Cela traduit le fait que la mise en place de la SPM en 2006 (correspond à la procréation du millésime 2007) a été concomitante à l'introduction de la sélection sur les caractères de morphologie de la mamelle (IMC). Pour la race Saanen, ces caractères sont en opposition génétique avec les caractères laitiers, donc plus difficiles à sélectionner conjointement. Pour la période SPMG, les premiers résultats semblent indiquer que le progrès génétique annuel sur l'ICC serait bien supérieur avec la génomique. Le gain annuel en race Saanen (+ 46 % par rapport à la période SPM) serait supérieur à celui en race Alpine (+ 29 %). Ces valeurs paraissent un peu surestimées au regard : i) des travaux de simulation qui avaient été faits en caprins avant le passage à la sélection génomique ; ii) des résultats obtenus dans d'autres espèces qui ont plus de recul en sélection génomique. Par exemple, Doublet et al. (2019), ont calculé un gain de progrès génétique annuel sur l'index de synthèse (ISU) de + 33 à +71 % entre sélection classique et génomique dans les trois principales races bovines laitières françaises. Dans les trois races ovines laitières des Pyrénées, qui ont des schémas de sélection de taille plus proche des caprins et une application de la sélection génomique récente, Astruc et al. (2022) montrent un gain de progrès génétique sur l'index de synthèse (ISOL) de +16 à +29 %.
Figure 4. Évolution de l’ICC (en unité d’écart-type génétique) par millésime de naissance des mâles d’IA en race Alpine et Saanen.
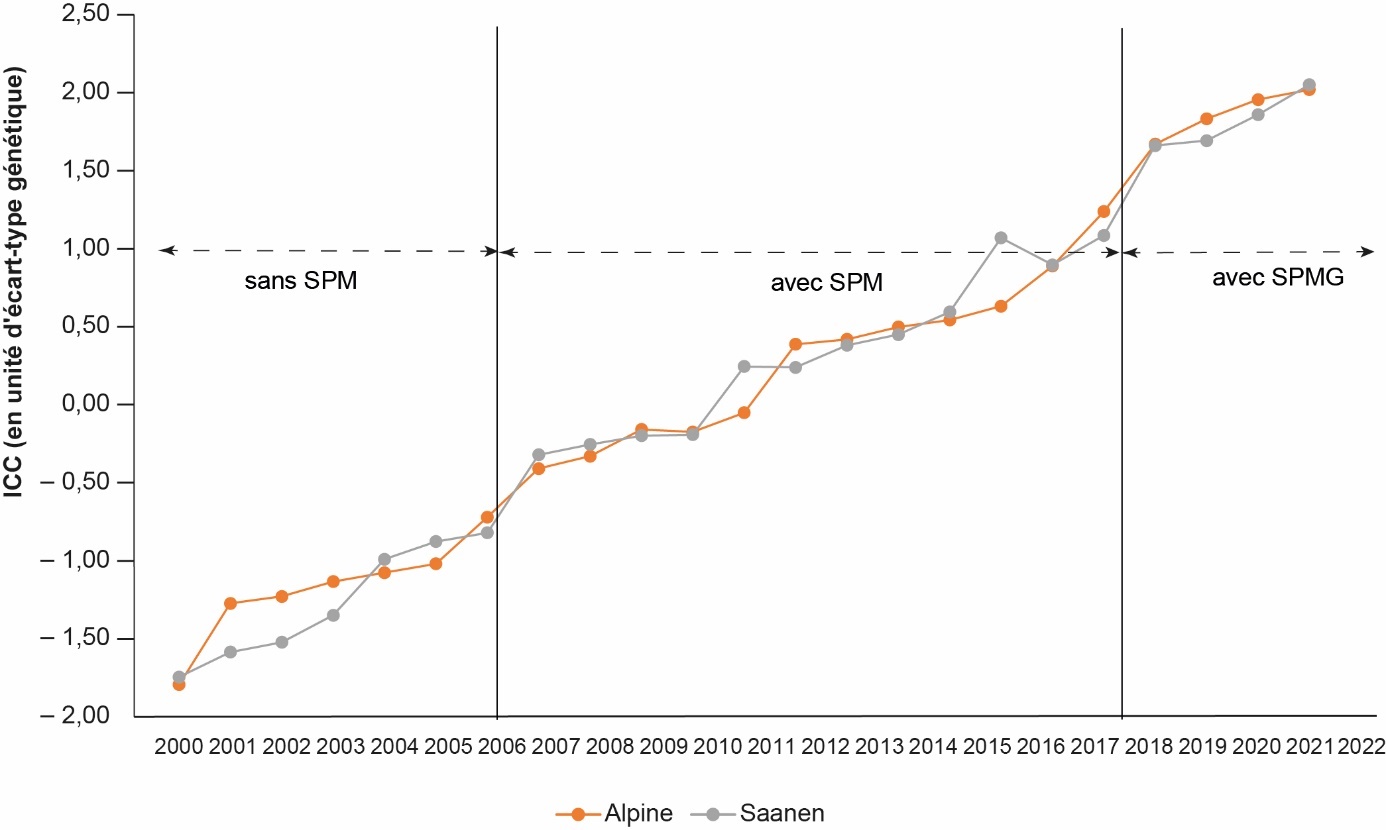
Les progrès génétiques sur l’IPC montrent les mêmes tendances que pour l’ICC (Tableau 7), de façon assez logique car l’IPC représente 55 à 60 % de l’objectif de sélection. L’IMC, introduit depuis 2006 en sélection, et l’index CCS, introduit depuis 2013, ont des progrès génétiques modestes (tableau 7), qui fluctuent beaucoup au cours des millésimes, sans tendance nette entre les périodes.
Il faut noter que la façon de regarder les choses ici, en considérant tous les males d’IA mis en testage, peut présenter un biais et n’est donc pas représentatif de ce qui se passe réellement à l’échelle du noyau de sélection. On considère en effet dans notre analyse que chaque mâle d’IA contribue de façon identique alors qu’une fois testé sur descendance, une sélection est opérée et seuls les meilleurs mâles continuent de faire des IA. La contribution aux IA de service est donc très différente selon les boucs (quelques centaines de doses à quelques milliers dans leur carrière). On peut penser que les mâles les mieux indexés sont plus utilisés. La réalité doit donc être un peu meilleure pour les progrès génétiques et un peu moins favorable pour la variabilité génétique que ce qui est décrit.
En résumé, il ressort que la mise en place de la SPM a été complètement bénéfique à la race Alpine pour laquelle les critères de variabilité génétique ont été fortement améliorés sans perte de progrès génétique sur l’index de synthèse global. Ceci est le reflet d’un niveau de variabilité génétique initial plus favorable et de l’absence d’une opposition entre caractères de morphologie et caractères laitiers. En race Saanen, la SPM semble un peu moins bénéfique car il y a une diminution du progrès génétique sur l’index de synthèse global et une amélioration moins forte des critères de variabilité génétique qu’en race Alpine. Toutefois, on peut supposer que la SPM a permis de limiter les dégâts inhérents à l’introduction des caractères de morphologie en sélection.
Même s’il est encore un peu tôt pour estimer l’effet de la SPMG avec précision, les tendances observées semblent indiquer que la génomique serait profitable à la fois sur la variabilité et le progrès génétiques et ceci, dans les deux races. La diversification des origines paternelles, générée par la SPMG et observée aujourd’hui sur les jeunes boucs mis en testage, devrait s’étendre petit à petit à toute la population, au cours des générations. On peut donc supposer que la variabilité génétique restera très satisfaisante à l’avenir.
4. Aperçu sur la diversité génétique des femelles du noyau de sélection
L’analyse des femelles du noyau de sélection apporte un regard intéressant et complémentaire à celui des mâles d’IA pour évaluer l’impact de la SPM (ou SPMG) sur la variabilité génétique. Elles sont en effet le reflet de l’utilisation réelle des mâles d’IA, chacun ayant contribué plus ou moins aux IA de service.
4.1. Sélection des femelles analysées et critères étudiés
Les femelles considérées sont les chèvres au contrôle laitier officiel, nées aux campagnes 2000 à 2020, de races Alpine ou Saanen et présentes dans un élevage ayant eu des pointages (seules les femelles du noyau de sélection sont pointées). Afin d’estimer des critères de variabilité génétique fiables, une sélection des femelles de père et grand-père maternel connus a été faite, soit 50 % des femelles en race Alpine et 43 % en race Saanen. Ainsi, l’analyse a porté sur 438 112 chèvres de race Alpine et 271 063 chèvres de race Saanen. Seules les consanguinités et parentés estimées à partir des pedigrees ont été considérées, compte tenu du très faible nombre de femelles génotypées. Puis les pentes de régression ont été calculées pour l’ensemble de la période 2000-2020.
4.2. Consanguinité et parenté des femelles
Le sous-ensemble des femelles de père et grand-père maternel connus présente une qualité des généalogies correcte et identique aux mâles d’IA avec, en moyenne, 9,5 équivalent générations remontées en race Alpine, 8,9 en race Saanen. L’augmentation annuelle de la consanguinité s’élève à 0,044 % en race Alpine et 0,059 % en race Saanen, ce qui est modéré et similaire aux résultats trouvés pour les mâles d’IA. Le taux d’augmentation de la parenté est, quant à lui, un peu plus élevé mais reste tout à fait satisfaisant : 0,068 % en race Alpine et 0,083 % en race Saanen. Ces résultats confirment que la gestion de la variabilité génétique appliquée pour la procréation des mâles d’IA via la SPM (ou SPMG) a un impact favorable sur l’ensemble du schéma de sélection.
Conclusion
Une méthode de type OCS (Optimal Contribution Selection) a été appliquée dans les schémas de sélection caprins des races Alpine et Saanen, à grande échelle et sur une durée conséquente (16 ans) en vue de la procréation des boucs d’IA. Dans le monde entier et pour l’ensemble des espèces domestiques animales, le nombre de programmes de sélection présentant ces caractéristiques est faible. L’étude présentée ici démontre l’efficacité d’une telle approche, à la fois pour générer du progrès génétique et préserver la variabilité génétique, même quand l’intervalle de génération est réduit grâce à une évaluation génomique précoce. Les deux schémas de sélection caprins ont en effet des critères de diversité parmi les meilleurs dans les populations de ruminants en sélection, tout au moins en France. Le progrès génétique annuel est également satisfaisant, quoiqu’il puisse être encore augmenté dans l’avenir avec les évolutions du schéma génomique projetées par l’organisme de sélection Capgenes. En effet, le taux global d’utilisation des boucs non testés sur descendance a été choisi de manière à être voisin du taux actuel pour l’insémination de service, c’est-à-dire 30 %. Les résultats obtenus montrent qu’il peut être quasiment doublé, en générant un progrès génétique supplémentaire, avec une augmentation encore raisonnable de la parenté et de la consanguinité. Il faut noter que les schémas de sélection caprins représentent une situation très favorable pour la mise en place efficace d’une telle méthode, en dépit de la taille somme toute assez faible de la population des reproducteurs utilisés en IA : i) la concurrence internationale est très faible donc il est plus facile de mettre l’accent sur la variabilité génétique ; ii) l’IA est faite avec de la semence congelée, ce qui permet une répartition aisée des doses des PAB sélectionnés par la SPMG dans tous les élevages sélectionneurs ; iii) la capacité reproductive d’un bouc d’IA est modérée car au plus 3 500 doses sont produites par bouc, limitant de fait la diffusion excessive de certains reproducteurs ; iv) les éleveurs sont tenus de respecter les accouplements programmés.
Dans le futur, la SPMG est amenée à évoluer. La prochaine étape vise à prendre en compte les génotypes pour certains gènes d’intérêt afin de contrôler l’évolution des fréquences à ces gènes, en plus des autres critères.
Références
- Astruc J.M., Baloche G., Buisson D., Labatut J., Lagriffoul G., Larroque H., Robert-Granié C., Legarra A., Barillet F., 2016. La sélection génomique des ovins laitiers en France. In : Brebis laitières en France : 50 ans de recherche et développement. Barillet F., Hassoun P., Astruc J.M., Lagriffoul G., Morin E. (Eds). INRA Prod. Anim. 29, 41-56. doi:10.20870/productions-animales.2016.29.1.2515
- Astruc J.M., Lagriffoul G., Legarra A., Buisson D., 2022, Six years of genomic selection have increased the genetic gain in French dairy sheep. 12th World Congr. Genet. Appl. Livest. Prod., Rotterdam, Pays Bas.
- Buisson D., Astruc J.M., Barillet F., 2018. Bilan et perspectives de la gestion de la variabilité génétique des ovins laitiers en France. INRA Prod. Anim., 31, 1-12. doi:10.20870/productions-animales.2018.31.1.2202
- Carillier C., Larroque H., Robert-Granié C., 2014. Comparison of joint versus purebred genomic evaluation in the French multi-breed dairy goat population. Genet. Sel. Evol., 46, 67. doi:10.1186/s12711-014-0067-3
- Carillier-Jacquin C., Larroque H., Robert-Granié C., 2017. Vers une sélection génomique chez les caprins laitiers. INRA Prod. Anim. 30, 19-30. doi:10.20870/productions-animales.2017.30.1.2228
- Chapuis H., Pincent C., Colleau J.J., 2016. Optimizing selection with several constraints in poultry breeding. J. Anim. Breed. Genet. 133, 1-10. doi:10.1111/jbg.12178
- Colleau J.J., Moureaux S., 2006. Gestion optimisée de la parenté et de la consanguinité dans les programmes de sélection des bovins laitiers. INRA Prod. Anim., 19, 3-14. doi:10.20870/productions-animales.2006.19.1.3476
- Colleau J.J., Tribout T., 2008. Optimized management of genetic variability in selected pig populations. J. Anim. Breed. Genet., 125, 291-300. doi:10.1111/j.1439-0388.2008.00738.x
- Colleau J.J., Moureaux S., Briend M., Béchu J., 2004. A method for the dynamic management of genetic variability in dairy cattle. Genet. Sel. Evol., 36, 373-394. doi:10.1186/1297-9686-36-4-373
- Colleau J.J, Tual K., de Preaumont H., Regaldo D., 2009. A mating method accounting for inbreeding and multi-trait selection in dairy cattle populations. Genet. Sel. Evol., 41, 7. doi:10.1186/1297-9686-41-7
- Colleau J.J., Fritz S., Guillaume F., Baur A., Dupassieux D., Boscher M.Y., Journaux L., Eggen A., Boichard D., 2015. Simulation des potentialités de la sélection génomique chez les bovins laitiers. INRA Prod. Anim., 28, 251-258. doi:10.20870/productions-animales.2015.28.3.3030
- Colleau J.J., Palhière I., Rodriguez Ramilo S.T., and Legarra A., 2017. A fast indirect method to compute genomic relationships concerning genotyped and ungenotyped individuals, for diversity management. Genet. Sel. Evol., 49, 487. doi:10.1186/s12711-017-0363-9
- Danchin-Burge C., Allain D., Clément V., Piacère A., Martin P., Palhière I., 2012. Genetic variability and French breeding programs of the three goats breeds under selection. Small Rum. Res., 108, 36-44. doi:10.1016/j.smallrumres.2012.03.016
- Doekes H.P., Veerkamp R.F., Bijma P., Hiemstra S.J., Windig J.J., 2018.Trends in genome-wide and regions specific genetic diversity in the Dutch-Flemish Holstein-Friesan breeding program from 1986 to 2015. Genet. Sel. Evol., 50, 15. doi:10.1186/s12711-018-0385-y
- Doublet A.C., Croiseau P., Fritz S., Michenet A., Hozé C., Danchin-Burge C., Laloë D., Restoux G., 2019. The impact of genomic selection on genetic diversity and genetic gain in three French dairy cattle breeds. Genet. Sel. Evol., 51, 52. doi:10.1186/s12711-019-0495-1
- Forutan M., Ansari Mahyari S., Baes C., Melzer N., Schenkel F.S., Sargolzaei M., 2018. Inbreeding and runs of homozygosity before and after genomic selection in North American Holstein cattle. BMC Genomics, 19, 98. doi:10.1186/s12864-018-4453-z
- Kirkpatrick S., Gelatt C.D., Vecchi M.P., 1983. Optimization by simulated annealing. Science, 220/671-680. doi:10.1126/science.220.4598.671
- Legarra A., Aguilar I., Misztal I., 2009. A relationship matrix including full pedigree and genomic information, J. Dairy Sci., 92, 4656-4663. doi:10.3168/jds.2009-2061
- Legarra A., Christensen O.F., Vitezica Z.G., Aguilar I., Misztal I., 2015. Ancestral relationships using metafounders: finite ancestral populations and across population relationships. Genetics, 200, 455-468. doi:10.1534/genetics.115.177014
- Metropolis N., Rosenbluth A.W., Rosenbluth M.N., Teller A.H., Teller E., 1953. Equations of state calculations by fast computing machines, J. Chem. Phys., 21, 1087-1092. doi:10.1063/1.1699114
- Meuwissen T.H.E, 1997. Maximizing the response of selection with a predefined rate of inbreeding. J. Anim. Sci., 75, 934-940. doi:10.2527/1997.754934x
- Palhière I., Clément V., Martin P., Colleau J.J., 2014. Bilan de la méthode de Sélection à Parenté Minimum après 6 ans d’application dans le schéma de sélection caprin. Renc. Rech. Ruminants, 21, 253-256
- Sonesson A.K., Woolliams J.A., Meuwissen T.H., 2012. Genomic selection requires genomic control of inbreeding. Genet. Sel. Evol., 44, 27. doi:10.1186/1297-9686-44-27
- VanRaden P.M., 2008. Efficient methods to compute genomic predictions, J. Dairy Sci., 91, 4414-4423. doi:10.3168/jds.2007-0980
- Villanueva B., Woolliams J.A., 1997. Optimization of breeding programmes under index selection and constrained inbreeding. Genet. Res., 69, 145-58. doi:10.1017/S0016672397002656
- Palhière I., Gousseau V., Colleau J.-J., 2002.La Sélection à Parenté Minimum Génomique : principes et résultats pour les deux races caprines laitières principales françaises. INRAE Prod. Anim., 35, 171-186.
Résumé
La Sélection à Parenté Minimum (SPM) est une stratégie de sélection performante d’optimisation combinée du progrès génétique et de la variabilité génétique des reproducteurs, développée en France au début des années 2000. Elle utilise la méthode dite du « recuit simulé ». Son application dans les schémas de sélection caprins a démarré en 2006 pour la procréation des mâles d’Insémination Artificielle (IA) par l’Organisme de Sélection (OS) français des deux races principales françaises, Alpine et Saanen. En 2018, alors que la sélection génomique est mise en place par l’OS dans les deux races, une version « génomique » de la SPM a été développée, tenant compte des informations génomiques pour évaluer les relations de parenté entre reproducteurs, et des différences de fiabilité des valeurs génétiques des boucs d’IA selon s’ils sont testés ou non sur descendance. Son efficacité est présentée pour les accouplements entre « pères à boucs » et « mères à boucs », réellement réalisés en 2021 par l’OS. Les animaux issus des accouplements présentent un niveau génétique global élevé), une consanguinité modérée (3,0 à 3,6 %) et peu de défauts sur les valeurs génétiques de chacun des caractères élémentaires en sélection. Le pourcentage de « pères à boucs » non testés sur descendance, critère clé nouvellement optimisé dans la SPM, est de 35 %. Il est atteint à la fois à l’échelle de tous les accouplements, et à l’échelle des troupeaux. L’étude des mâles d’IA nés entre 2000 et 2020 permet de mesurer l’impact réel de la SPM sur le long terme et d’estimer les premiers effets de la version génomique. Le progrès génétique annuel sur l’index de synthèse correspondant à l’objectif de sélection est conséquent avec 0,17 écart-type génétique dans chacune des races. En parallèle, le taux d’augmentation de la consanguinité par génération est très modéré : 0,26 % en race Alpine et 0,21 % en race Saanen.
Pièces jointes
Pas de document complémentaire pour cet articleStatistiques de l'article
 Vues: 2138
Vues: 2138
Téléchargements
 PDF: 466
PDF: 466
 XML: 171
XML: 171

